Nature:清华大学施一公发表最新论文
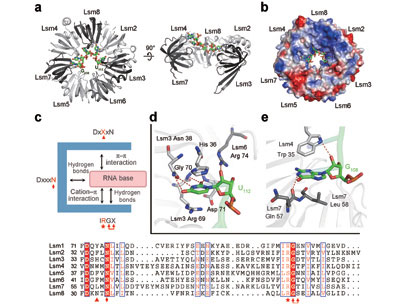
清华大学施一公研究组在《自然》发表最新论文,阐述Lsm蛋白质复合物特异性识别剪切体U6 RNA的分子机制。图示Lsm2-8复合物与U6 RNA的特异性识别。
11月17日,清华大学生命学院施一公教授研究组在国际顶级学术期刊《自然》在线发表了题目为CrystalstructuresoftheLsmcomplexboundtothe3’endsequenceofU6smallnuclearRNA(Lsm蛋白质复合体结合U6小核RNA3’末端序列的晶体结构)的研究论文,首次报道了Lsm2-8蛋白质复合物自组装的晶体结构及其特异识别U6小核RNA3’末端序列的分子机制。清华大学生命学院博士生周丽君和医学院博士生杭婧为该论文的共同第一作者。
在真核生物中,执行翻译功能的信使RNA(messengerRNA)在细胞核内的成熟需要经历一个非常复杂的剪切和拼接过程,这个过程主要是由五个小核核糖核蛋白(smallnuclearribonucleoproteins,snRNP)和一系列的辅助蛋白构成的巨大分子机器——剪切体(spliceosome)执行的。每种snRNP都由一条小核RNA(smallnuclearRNA)和与之特异性结合的七元环蛋白质复合物组成。大部分的七元环复合物是Sm蛋白质七聚体,只有在U6小核核糖核蛋白中为Lsm蛋白质七聚体复合物。Lsm蛋白家族有十多个成员,在各个物种中高度保守,参与各种与RNA代谢相关的信号通路。在真核细胞中,组成U6小核核糖核蛋白的蛋白质复合物是Lsm2/3/4/5/6/7/8。它能够特异地识别U6RNA的3’末端序列,并帮助U6RNA与剪切体其他成员相互作用,催化RNA的剪接过程。
在这篇研究论文中,施一公研究组通过特殊的蛋白质表达手段获得了均一性和稳定性良好的自组装Lsm蛋白质复合物,克服了传统Lsm蛋白提取方法对蛋白质造成的潜在变性危害。在此基础上,作者对蛋白质复合物和RNA片段进行了共结晶,结合结构生物学和生物化学的分析手段,揭示了Lsm2-8七聚体蛋白质复合物特异性识别U6snRNA末端的分子机制。Lsm2-8蛋白质复合物中的四个亚基Lsm4/8/2/3分别使用两个保守的序列模式特异性地把U6RNA3’末端4个尿嘧啶锚定在七聚体圆环形成的中间空腔内,其中Lsm3识别最末位的尿嘧啶核苷酸。有趣的是,当这个核苷酸被截去之后,Lsm3仍可以以及其相似的识别模式锚定最后一个尿嘧啶,引起整个结合序列的后移。除与Lsm4相邻的Lsm7对RNA的结合贡献了微弱的氢键以外,其他两个亚基并没有直接参与到RNA识别中去。这一创造性的新发现首次从三维晶体结构上描述和解释了Lsm蛋白对U6RNA的“末端识别”模式。
由于剪接通路的动态复杂性,自1993年基因剪接的发现被授予了诺贝尔生理学医学奖以来,科学家们仍在步履维艰地探索着其中的奥秘。此课题的完成攻克了世界上多个研究组感兴趣的难题,将7个不同蛋白质共同表达并结晶。为此领域的相关研究提供了新的设计思路和研究方法。
Lsm蛋白质复合物晶体结构的解析是施一公研究组首次在RNA剪接通路中取得的重大进展,为更好地理解真核生物剪接体的功能实现和揭示生命现象的基本原理奠定了夯实的理论基础。

