PLoS Genetics:美科学家发现粘细菌识别相关菌株分子机制
美国怀俄明州立大学科学家的一项最新研究报道,他们发现了粘细菌识别相关菌株的分子机制,这种机制能引起其外膜短暂融合以交换脂肪和蛋白。此项研究在11月7日的《PLOS genetics》杂志上发表。
美科学家发现粘细菌识别相关菌株分子机制
研究发现,黄色粘球菌交换细胞成分来帮助单个细胞转变成多细胞生命。而且,这个机制在细菌如何适应压力——包括抗生素耐药性中发挥作用。
单个细胞如何识别彼此、合作和组装成功能性组织是生物学界的一个根本问题。尽管多细胞性是真核生物的一个典型特征,某些群体的细菌也表现出复杂的多细胞行为,粘细菌就是一个最好的例子。例如,在饥饿条件下,粘细菌会聚集形成子实体,在子实体中,数以千计的细胞在发育和细胞变异中起一个“连贯单元”的作用。然而,粘细菌或者其它细菌,是如何通过细胞接触-依赖相互作用来识别合作的搭档细胞,仍然没有充分的了解。
粘细菌识别机制
由美国怀俄明州立大学农业和资环学院生物分子系的副教授Daniel Wall带领的团队,描述了一种机制,通过这种机制,粘细菌辨别兄弟姐妹和来自其它粘细菌的同生群细胞。研究表明,这种分子识别是由一个称为TraA的细胞表面受体介导的。
黄色粘球菌是一种土壤细菌,Wall称,这种细菌非常的与众不同,他们是有社会性的。它们彼此之间相互作用,组成细胞单元,表现出例如越过土壤表面的团体运动这样的行为。在饥饿条件下,近缘菌也会聚集形成成多细胞结构。Wall说,“它们生活在土壤中,这是一种由成千上万不同微生物种类组成的世界性环境,粘细菌具有识别彼此的能力,在饥饿条件下,它们聚集在一起,建立肉眼可见的惊人的子实体结构”。这些结构中的一些,具有类似树一样的茎和分枝。细菌的聚集使它们离开一个表面,能够被风、水或者动物传播到一个更可持续的环境。
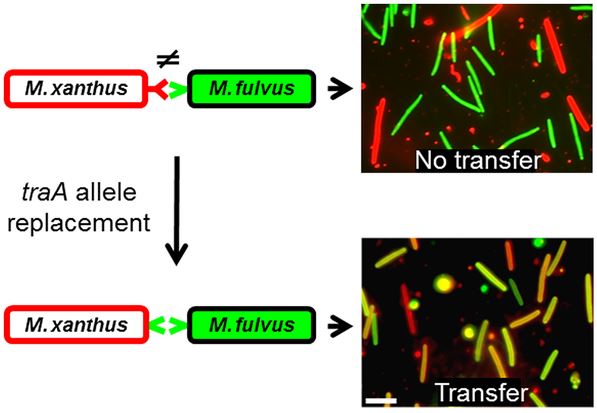
Wall想确定细胞识别的分子机制。他们断定,traA——一个外膜物质传送所必需的基因,起了一个细胞表面受体的作用。他们也发现,粘细菌在它们合伙进行外膜交换时,表现出具有歧视性的行为,这些有选择性的相互作用与在周围病原体的TraA受体中发现的超变量序列变异有关。
为了验证是否TraA多态性控制细胞间的识别,研究者将traA等位基因从一个菌株交换到另外一个,发现产生的菌株会识别属于新traA等位基因的群组,而不是其旧的识别群组。Wall称,“这个实验证明,traA是分子特异性的决定因素”。
细菌间为什么发生交换
Wall说:“我们没有完整的答案,我们认为它们分享细胞物质,来构建一个细胞的合作团队,这个团队能够在多细胞活动中发挥作用。目前,我们了解到,没有其它细菌统表现这种行为——细胞具有涉及识别的一个受体,引起膜融合和物质交换。”
研究者推测,这种交换是细菌修复受损细胞的一种方式。搭档细胞在近似相等的水平分享可交换的物质。如果一个细胞缺乏一个特定蛋白,经过外膜交换后,交换的两个细胞将具有相同数量的这种蛋白。
Wall说,“当你想一想,细菌生活在它们的环境中,不同年龄的细胞,接触到各种各样的环境损害和捕食者,因此细胞群体就变的多样化——有些细胞是健康的,有些是受损伤的,其它的可能是饥饿的。外膜交换使得细胞与细胞因子混合,我们推测这将使种群变得更均匀和更健康” 。
Wall称,相同种类的细菌——而不是不同的菌株或病原体,彼此之间通常是有对抗性的。“人们经常可以发现,一个菌株会杀死相同种类的另外一个菌株。”
Wall说,这些成分通常被称为细菌素——特殊的肽类或者蛋白,能够识别其它亲缘关系很近的细菌,除了编码一个免疫蛋白之外,还能杀死它们。产生细菌素的细胞也不得不产生一个免疫因子保护它自己免受毒素的伤害。任何不具有这个免疫因子的相关邻近细胞都会被杀死。他说,“在两个不属于同一识别组群的菌株之间交换traA基因,使它们能够交换外膜物质,为它们提供保护,使它们不被杀死。我们已经解释了这个结果,意味着有一个免疫因子能够在细胞间转运”。
Wall称,引申开来,交换过程也在抗生素抗性中发挥作用。这项工作为细菌细胞如何相互作用描述了一种新典范,也对微生物在我们体内是如何相互作用提出了新的见解。“细胞能够利用这种机制进行沟通交流,新功能能在细胞间转运,”他说。
这项研究也适用于发生在细菌的地盘——土壤中的过程。“我们实验室的粘细菌物种在土壤中很常见,”Wall说。“它们捕食其它细菌和真菌生物,包括农作物病原菌。粘细菌也不会伤害作物,在一个单独的项目中,我们正在探索粘细菌作为作物保护的一种生物控制剂的实用性。
原文摘要:
Molecular Recognition by a Polymorphic Cell Surface Receptor Governs Cooperative Behaviors in Bacteria
Darshankumar T. Pathak equal contributor, Xueming Wei equal contributor, Arup Dey, Daniel Wall
Cell-cell recognition is a fundamental process that allows cells to coordinate multicellular behaviors. Some microbes, such as myxobacteria, build multicellular fruiting bodies from free-living cells. However, how bacterial cells recognize each other by contact is poorly understood. Here we show that myxobacteria engage in recognition through interactions between TraA cell surface receptors, which leads to the fusion and exchange of outer membrane (OM) components. OM exchange is shown to be selective among 17 environmental isolates, as exchange partners parsed into five major recognition groups. TraA is the determinant of molecular specificity because: (i) exchange partners correlated with sequence conservation within its polymorphic PA14-like domain and (ii) traA allele replacements predictably changed partner specificity. Swapping traA alleles also reprogrammed social interactions among strains, including the regulation of motility and conferred immunity from inter-strain killing. We suggest that TraA helps guide the transition of single cells into a coherent bacterial community, by a proposed mechanism that is analogous to mitochondrial fusion and fission cycling that mixes contents to establish a homogenous population. In evolutionary terms, traA functions as a rare greenbeard gene that recognizes others that bear the same allele to confer beneficial treatment.

