Nucl Acids Res:北京大学孙育杰组开发基于改造sgRNA的活细胞染色质
2016年2月4日,国际知名核酸研究期刊《Nucleic Acids Research》杂志在线发表北京大学生命科学学院生物动态光学成像中心孙育杰课题组题为“Long-term Dual-color Tracking of Genomic Loci by Modified sgRNAs of the CRISPR/Cas9 System”的研究文章。文章报道利用可编辑的内切酶失活的人工核酸酶(CRISPR/dCas9)作为DNA结合蛋白,开发了一种新的基于改造的功能性扩展sgRNA的CRISPR多色、稳定的荧光成像系统。北京大学三年级博士生邵世鹏是该论文的第一作者,北京大学生物动态光学成像中心孙育杰研究员是该论文的通讯作者。
基因在细胞核中的位置和细胞生命活动中基因位点的动态变化是真核细胞基因表达调控的一个重要层面。因此对活细胞内染色体基因位点运动过程实现高效,可信,稳定的多色多基因位点同时标记观测与追踪对于理解拥挤细胞核内很多动态过程非常关键。近几十年已经发展了多种DNA标记技术,其中最近出现的利用可编辑的DNA结合蛋白进行标记的方法因操作方便、可扩展性好等优点脱颖而出。
CRISPR系统的特异性是由sgRNA决定而非Cas9蛋白,传统的利用dCas9直接标记成像只能实现单色标记。该工作开发了基于改造sgRNA的基因位点多色标记新方法。当改造的sgRNA与dCas9形成复合物时,可以结合到靶位点并且通过适配子结合蛋白招募荧光蛋白到靶位点进行放大。文章通过对染色体端粒和着丝粒同时成像证明了标记系统在双色成像上的表现。通过与传统标记方法的共定位说明了新开发系统的特异性。与传统的荧光原位杂交(FISH)和多重重复片段插入(FROS)等技术相比,这种新的成像方法适合标记活细胞,操作简单、易实现。与dCas9标记方法相比,基于改造sgRNA的方法提供了更加快速交换的动力学,可以抵抗光漂白的影响,特别适合对于基因位点的长时间连续观察追踪。基于改造sgRNA的多色成像方法具有较高的特异性、信噪比、灵敏度、通用性,可以和基于正交Cas9的多色成像方法进行互补,进一步提高多色标记的通量。该技术未来可以应用于多基因的共转录,DNA修复,染色质的结构变化等方面的研究,对于理解基因位置与基因表达调控具有重要的意义。
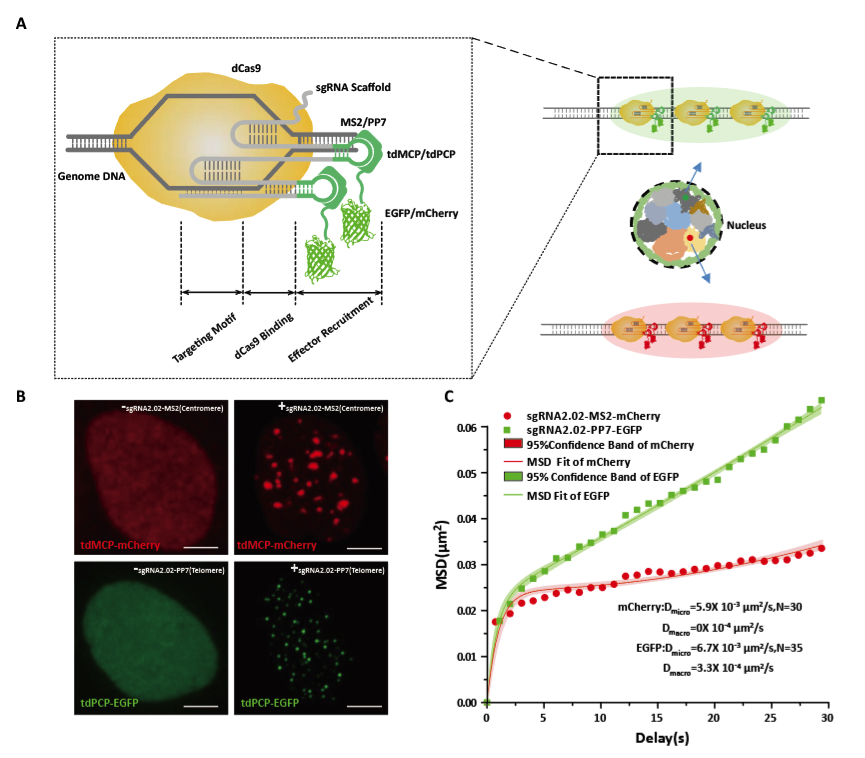
改造sgRNA的原理示意图和标记效果图
原文链接:
Long-term dual-color tracking of genomic loci by modified sgRNAs of the CRISPR/Cas9 system
原文摘要:
Visualization of chromosomal dynamics is important for understanding many fundamental intra-nuclear processes. Efficient and reliable live-cell multicolor labeling of chromosomal loci can realize this goal. However, the current methods are constrained mainly by insufficient labeling throughput, efficiency, flexibility as well as photostability. Here we have developed a new approach to realize dual-color chromosomal loci imaging based on a modified single-guide RNA (sgRNA) of the CRISPR/Cas9 system. The modification of sgRNA was optimized by structure-guided engineering of the original sgRNA, consisting of RNA aptamer insertions that bind fluorescent protein-tagged effectors. By labeling and tracking telomeres, centromeres and genomic loci, we demonstrate that the new approach is easy to implement and enables robust dual-color imaging of genomic elements. Importantly, our data also indicate that the fast exchange rate of RNA aptamer binding effectors makes our sgRNA-based labeling method much more tolerant to photobleaching than the Cas9-based labeling method. This is crucial for continuous, long-term tracking of chromosomal dynamics. Lastly, as our method is complementary to other live-cell genomic labeling systems, it is therefore possible to combine them into a plentiful palette for the study of native chromatin organization and genome ultrastructure dynamics in living cells.
作者:孙育杰

