物理所原核生物染色体DNA自组装模式和机理研究获进展
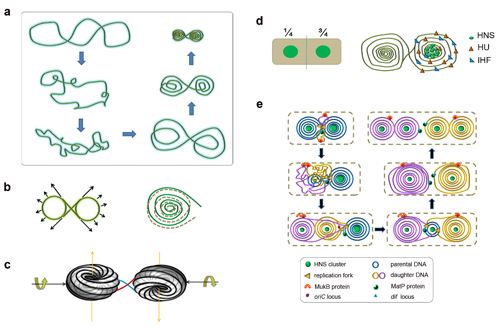
原核生物大肠杆菌染色体DNA体内盘绕方式模型示意图。(a)质粒DNA从负超螺旋构象演化成“双重螺线圈”构象的二维路径示意图。(b)“双重螺线圈”构象具有自对称“8”字形骨架(左)和两个由外入内渐进盘绕的螺线圈(右)。(c)大肠杆菌染色体DNA体内静态盘绕的三维立体模式
生命机体内部有机生物大分子种类构造极其繁复,其运转却呈现出高度有序、协调和精妙的自我调控。在纷繁复杂的生物反应中,染色体DNA自组装、复制和应时启动转录处于细胞生命活动的中心位置。虽然人类基因组计划已经完成,但是数以亿计的DNA遗传信息凝聚在十几微米大小的细胞核内,目前,我们还没有能力清晰理解这些信息是如何被细胞利用、提取、整合并最终发布成效应蛋白质来应对正常生命活动和细胞内外信号刺激。这些基本调控运转机制正是攻克人类疾病和药物医疗研发的必需前提。为了从根本上理解生命运作本质,避繁就简,基础研究生物学家们几十年以来一直以原核生物——最具代表性的大肠杆菌等为研究模型,建立了一整套成熟高效的基因蛋白操纵手段,然后力求把原核细胞中的发现借鉴到真核生物及最终目标人类细胞之中,取得了卓有成效的进展。令人惊讶的是:迄今为止,原核生物细菌染色体DNA组装方式仍是未解之谜。人们早在几十年以前已经用电镜观察到真核生物的染色质DNA和组蛋白构成的“串珠”模式核小体,病毒的基因组DNA是紧密排布于衣壳内。相比之下,生物界另一大类原核生物,人们对其DNA组装方式至今仍未能窥其一瞥,归因于实验技术手段的限制,使得生物学家们如此熟悉的经典研究模型细菌,又是如此神秘和陌生。
最近,中国科学院物理研究所/北京凝聚态物理国家实验室(筹)软物质物理重点实验室SM1组副研究员刘玉如、研究员谢平、研究员王鹏业和SM4组研究员李明等对大肠杆菌染色体DNA组装进行了理论和实验研究。先前王鹏业和刘玉如在研究铂类抗癌药物与DNA单分子作用机理时,意外地观察到了环形质粒DNA在铂类药物协同作用下自发盘绕成“双重螺线圈”的奇异现象。超螺旋环形DNA分子在局部弯折的诱导下,一律在溶液中自发形成自我对称的8字形“双重螺线圈”构象,并通过原子力显微镜成功地观察到其动态演变过程。借助于近年来该研究组深入进行DNA大分子对力和铂类药物作用响应生物物理特性研究的积累,他们对这一奇异现象发生的原理进行了解释和推演。由于DNA大分子和铂类药物反应过程的复杂性,通过理论计算DNA局部形变和DNA分子内部能量进行分子动力学模拟在目前还做不到。但是,他们从最基本的物理规律入手,创新性地提出了支配DNA大分子自发盘绕的动态平衡和能量最低原理。
受到上述的体外环型质粒DNA在顺铂和反铂作用下构型变化的启发,考虑到原核生物的染色体DNA也是超螺旋环形DNA,刘玉如等进一步提出了大肠杆菌染色体DNA在活体内一个复制周期中各个阶段的盘绕状态以及支配其自组装的理论模型。利用软物质物理重点实验室蛋白质表达公共实验平台,他们成功构建了基因原位可激发荧光蛋白标记的HNS-mEos2大肠杆菌株,通过PALM超高分辨率荧光显微成像技术,对不断分裂大肠杆菌HNS-mEos2荧光蛋白簇的演化进行了实时动态观测。HNS是一种类核相关蛋白,其伴随着DNA的凝聚,可用其来示踪DNA盘绕状态。他们实验中明确观察到了HNS在新生大肠杆菌中形成两个对称荧光团簇,随着DNA的复制,团簇先是变模糊,然后再在预分裂细胞中形成明确的四个团簇。实验结果与DNA“双重螺线圈”理论模型完全吻合,每一个HNS荧光团簇指示出了一个DNA“螺线圈”。这一模型也支持了细菌染色体凝聚在类核中是内在力学性质扮演支配角色这一观点。同时,该模型可以很好地解释此研究领域中绝大部分以往观测到的实验结果,并且统一了明显矛盾的实验现象。该研究在国际上首次提出原核生物大肠杆菌染色体DNA具体三维的组装盘绕模式,以新的理论和视角分析了染色体DNA在细胞内狭小空间内高度有序自组装的潜在机理,深入了人们对生物体最基本的染色体DNA自组装现象的理解。鉴于目前科学技术手段的局限,对原核生物染色体DNA分布的直接观测还不可能,因而对于此理论模型的直接观测证据还有待于未来研究提供。该项研究结果于11月24日发表在Scientific Reports(doi: 10.1038/srep17133 (2015))。
该工作得到了科技部“973”项目(课题号:2013CB837200)、国家自然科学基金项目(课题号:11204363,11274374,61275192)和中国科学院的支持。

