The Plant Journal:南京大学生科院田大成课题组发表水稻稻瘟病研究
2015年10月1日植物学著名杂志《The Plant Journal》上在线发表南京大学生命科学学院田大成教授课题组在水稻稻瘟病抗性基因的研究中取得的重要进展,研究成果题为“A genome-wide survey reveals abundant rice blast R genes in resistant cultivars”。论文的第一作者为南京大学生命科学学院张小辉博士,第一通讯作者是田大成教授。研究得到南京大学仙林校区作物分子遗传实验站的支持。
水稻稻瘟病是水稻最重要的三大病害之一。稻瘟病菌是一种变异与分化速度快、多样性程度高、新的生理小种层出不穷的水稻病害。长期的生产实践证明,抗病育种(利用自身的抗病基因)是防治稻瘟病最经济有效的措施,也符合人类对绿色食物的要求。传统的图位克隆抗病基因的方法虽然取得了比较好的成效,但整个克隆过程周期长,效率不高。
为此,田大成课题组另辟新径,建立了以植物与病菌共进化为基础的基因克隆方法,通过各种遗传参数的计算来挑选候选基因,并通过遗传转化、抗性鉴定等环节,共获得98个功能性抗稻瘟病基因,其中含15个抗谱广、抗性强的抗稻瘟病基因。
抗病基因克隆技术的突破,具有重要科学意义:
1)建立了一种全新的植物抗病基因高通量克隆方法,大大提高了抗病基因的克隆效率,为克隆更多更好的广谱高抗基因奠定了基础;
2)首次从全基因组系统评估了抗稻瘟病基因的功能与分布,为后续植物抗病机制研究奠定了坚实的基础;
3)克隆的广谱高抗基因的,可以直接应用于主要作物的抗病育种,具有重大的应用价值;
4)为其它植物抗病基因的高效克隆提供了可行的操作范例和技术指导。
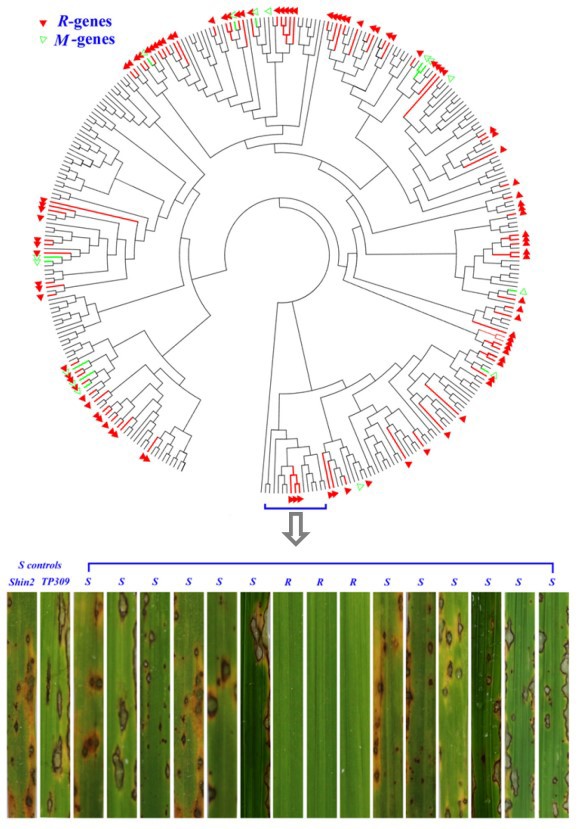
图:克隆的332个NBS-LRR基因的进化关系和转基因株系稻瘟病菌侵染后的代表表型。
原文链接:
A genome-wide survey reveals abundant rice blast R genes in resistant cultivars
原文摘要:
Plant resistance genes (R genes) harbor tremendous allelic diversity, constituting a robust immune system effective against microbial pathogens. Nevertheless, few functional R genes have been identified for even the best-studied pathosystems. Does this limited repertoire reflect specificity, with most R genes having been defeated by former pests, or do plants harbor a rich diversity of functional R genes, the composite behavior of which is yet to be characterized? Here, we survey 332NBS-LRR genes cloned from five resistant Oryza sativa (rice) cultivars for their ability to confer recognition of 12 rice blast isolates when transformed into susceptible cultivars. Our survey reveals that 48.5% of the 132 NBS-LRR loci tested contain functional rice blast R genes, with most R genes deriving from multi-copy clades containing especially diversified loci. Each R gene recognized, on average, 2.42 of the 12 isolates screened. The abundant R genes identified in resistant genomes provide extraordinary redundancy in the ability of host genotypes to recognize particular isolates. If the same is true for other pathogens, many extant NBS-LRR genes retain functionality. Our success at identifying rice blast R genes also validates a highly efficient cloning and screening strategy.
作者:田大成

