微生物所在链霉菌合成生物学调控元件定量分析方面获得新进展
链霉菌是抗菌药物、农药、抗癌药物和免疫抑制剂等具有生物活性的微生物次级代谢产物的一个重要来源,但令人沮丧的是,利用传统的高通量筛选方法和单菌多产物(one strain many compounds)策略发现新药物的难度越来越大。链霉菌基因组的破译展示给我们大量未被解析的基因簇,预示着新的生物活性物质的存在。尽管链霉菌的基因组比酿酒酵母小得多,却具有更多的ORF,大部分用于调控基因簇的转录和翻译。然而由于链霉菌丝状生长的特性使其基因表达调控难以精确设计、定量和预测,成为合成生物学在该领域发展的一个巨大的技术瓶颈。
最近,一项发表在《美国科学院院报》上的研究结果为我们解决了上述难题。来自中国科学院微生物所的娄春波研究员和张立新研究员的研究团队合作建立了一个基于流式细胞仪和绿色荧光蛋白sfGFP的单细胞精确定量方法,用于鉴定链霉菌中的调控元件。
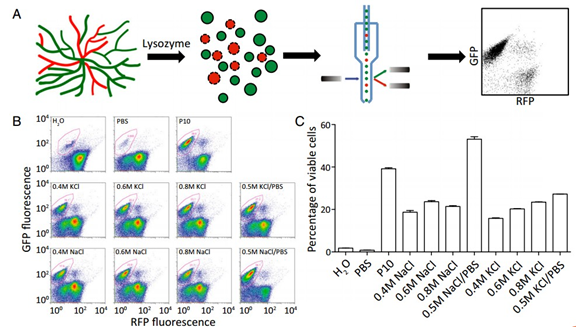
他们首先在链霉菌基因组中引入强启动子表达的sfGFP基因,然后利用溶菌酶制备原生质体。由于制备原生质体过程中保持原生质体渗透压的蔗糖缓冲液会损坏流式细胞仪,他们使用了另一种缓冲液替代了蔗糖缓冲液,同时使用碘化丙啶(propidium iodide ,PI)染色区分死细胞,然后利用共焦激光扫描荧光电子显微镜扫描。利用该方法,他们对200个天然的或合成的启动子和200个RBS进行高通量表征。另外又利用绝缘子RiboJ消除了启动子和RBS之间的干涉,提高了这些调控元件的模块性。利用这些调控元件,他们以一种可预测的方式成功地激活并提高了阿维链霉菌(Streptomyces avermitilis)中的沉默基因簇番茄红素的表达。他们的研究提供了一个定量测量方法以及通用的模块化调控元件,促进了链霉菌中基因簇功能优化和药物发现过程。
段春兰 摘译自: http://www.pnas.org/content/early/2015/09/14/1511027112
更多生物催化内容请关注微信公众服务号:生物催化剂设计与改造服务(STS-iDeep)

(发布:)