OMICS:中科院北京基因组所于军与伦敦大学王大鹏合作开发基因
基因在染色体上的排布(Gene Orders)是高度组织化的,在自然选择作用下,单一基因的结构和成簇基因的排列顺序在不同进化分支中具有不同程度的可变性,并且基因排布与染色体结构的关系仍然有待进一步探索。进化基因组学的重要研究内容之一就是揭示在这些大规模且不同程度的基因排布下,不同物种基因组在基因共调控机制方面的相似和差异。现今,二代测序技术的快速发展和广泛应用,产生了不同分类群代表物种的基因组数据,但是缺乏一个基于大规模基因组数据且操作简单的基因排布可视化工具。
为此,中国科学院北京基因组研究所基因组科学与信息重点实验室研究员于军和英国伦敦大学学院肿瘤研究所王大鹏合作开发了一套全新的网络可视化工具LCGserver,用于解析多类群进化背景下基因排布的动态变化规律,此项研究于近期发表在OMICS: A Journal of Integrative biology 杂志。
针对基因排布,LCGserver提供三个水平的分析功能:单个基因、成对基因和成簇基因。其独特的功能是可以根据成组基因的同源性将不同的基因组进行比对,帮助用户直观地鉴定特定物种基因组在特定进化分支上基因排布的保守和变异事件。另外,根据保守和变异的所有可能性,将成对基因分成6种模式并且可以识别完全保守的基因簇的存在。
此项研究将对进化和比较基因组学领域产生积极影响,比如研究新测序物种基因组和公共数据库基因组的基因共线性关系,通过比较近缘物种基因组查看并纠正组装过程中存在的问题,通过基因位置信息构建物种进化树,与rna-seq数据结合进行基因位置和基因共表达的交叉验证,高度保守的基因簇可以作为高维基因组结构的锚点等。
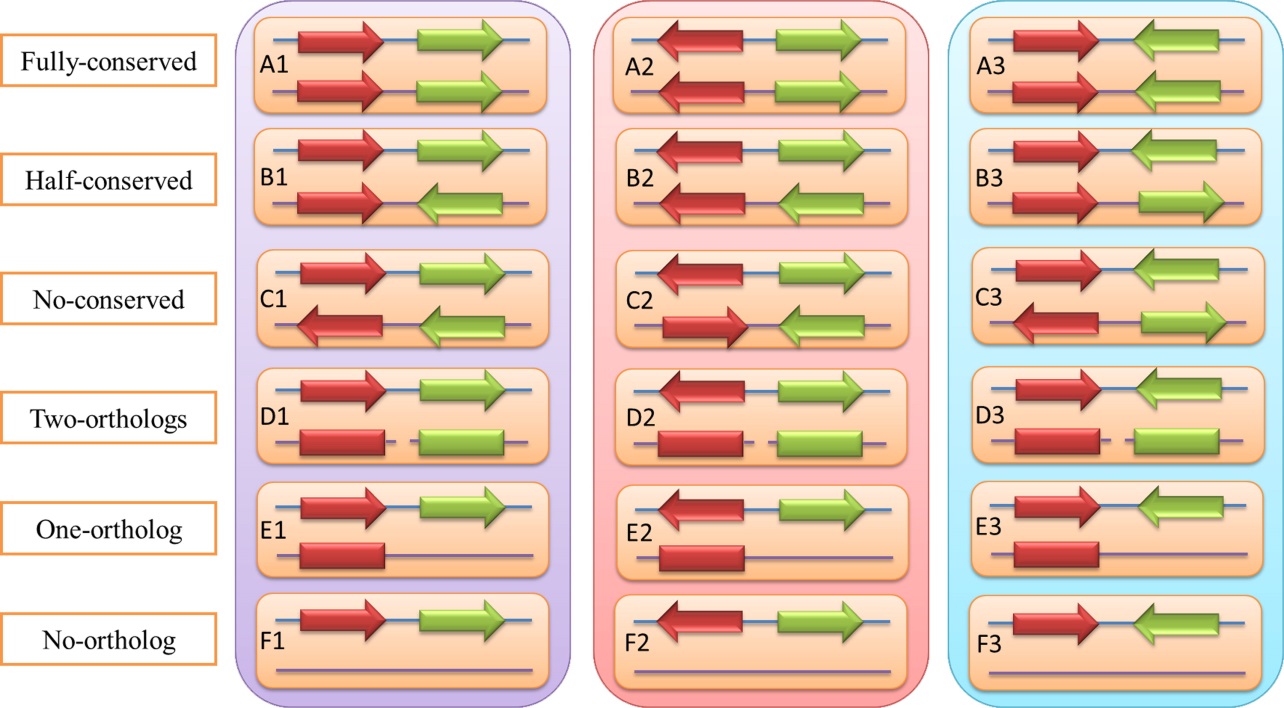
成对基因的六种变异模式
原文链接:
LCGserver: A Webserver for Exploring evolutionary Trajectory of Gene Orders in a Large Number of Genomes
原文摘要:
Genes and chromosomes are highly organized; together with protein-coding sequence, gene structure at per gene level and gene order at cluster level are both variable in a context of lineages and under natural selection. How gene order and chromosome organization are related and selected remains to be Illuminated. The number of newly-sequenced genomes from various taxa has been increasing rapidly, but there have not been easy-to-use web tools that allow better visualization for gene order in a large genome collection. Here, we describe a webserver, LCGserver (http://lcgbase.big.ac.cn/LCGserver/), for exploring evolutionary dynamics of gene orders over diverse lineages. This server provides gene order information at three levels: single gene, paired gene (a minimal cluster), and clustered gene (more than two genes). The most exclusive feature of LCGserver is alignment and visualization of neighboring genes based on orthology, allowing users to inspect all conserved and dynamic events of gene order along chromosomes in a lineage-specific manner. In addition, it categories paired genes into six patterns and identifies fully-conserved gene clusters within and among lineages.
doi:10.1089/omi.2015.0060.
作者:于军与王大鹏

