我国科学家用CRISPR实现杨树定向诱变

最近,RNA引导的CRISPR/Cas基因组编辑,已被用于多种草本植物的基因组编辑。然而,该系统是否可用于木本植物的基因组编辑,仍然还是非常罕见的。今年六月份,美国乔治亚大学(UGA)的研究人员首次利用CRISPR/Cas基因编辑工具,修改杨属植物的基因组。他们的研究结果发表在植物学国际知名期刊《New Phytologist》,为更快速和更可靠的植物基因编辑,打开了大门。相关新闻:首次用CRISPR编辑树木基因组。
七月二十日,来自西南大学、中科院西北高原生物研究所的科学家,在Nature子刊《Scientific Reports》发表的一项研究中,通过CRISPR/Cas系统实现林木植物毛白杨(Populus tomentosa Carr.)的基因组编辑和靶基因突变。
本文通讯作者为西南大学生命科学学院教授、博士生导师罗克明,其1999年毕业于西南师范大学生物系,1999年9月至2004年7月,为西南农业大学与美国康涅狄格大学联合培养博士,2001年12月至2003年10月,美国康涅狄格大学访问学者,2005年2月至2007年2月,西南大学生物技术中心副研究员,2007年3月至2008年5月,在美国密歇根理工大学从事博士后研究,2008年7月至今,西南大学生命科学学院教授。教育部“新世纪优秀人才计划”获得者,中科院“百人计划”入选者。主要研究方向:三峡库区特色资源植物功能基因研究及基因工程;转基因植物生物安全性评估、检测和控制技术的研究,曾在Molecular Biology、Plant Cell Reports、PLoS ONE、J. Exp. Bot.等学术期刊发表论文多篇。
作为一个最广泛种植的速生树种,杨树具有巨大的经济价值和生态价值。自从2006年毛果杨(Populus trichocarpa)全基因组序列发布以来,现在有广泛的基因组资源可用于这个树种的功能基因组学研究,它已被用作森林遗传学和木本植物研究的一个模型。因此,了解杨树基因功能和转录调控的分子机制,对于树木遗传工程和可持续的森林管理,是至关重要的。然而,相比较拟南芥、水稻和其他的一年生模式植物,木本植物由于营养生长期较长、遗传转化效率低和突变体数量有限,更难进行功能基因组学研究。尽管一些有前途的方法,可以产生杨树的基因敲除突变体,但是到目前为止,仍然缺乏大规模的基因突变体资源。
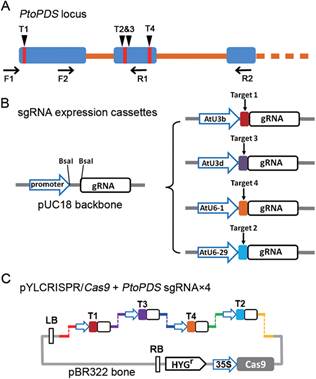
在这项研究中,研究人员利用CRISPR/Cas9系统,在毛白杨中实现了基因组编辑和靶基因突变。研究人员设计了四个引导RNA(gRNA),来靶定毛白杨八氢番茄红素脱氢酶基因8 (PtoPDS)的不同基因组位点。在农杆菌介导的转化之后,研究人员在转基因杨树中观察到了明显的白化表型。通过分析RNA引导的基因组编辑事件,59个PCR克隆中有30个是纯合子突变体,有2个是杂合子突变体,这些目标位点的突变效率估计是51.7%。这些数据表明,我们可以利用Cas9/sgRNA系统,在木本植物中精确地编辑基因组序列,并有效地建立基因敲除突变体。(来源:生物通 王英)
Efficient CRISPR/Cas9-mediated Targeted Mutagenesis in Populus in the First Generation
Abstract Recently, RNA-guided genome editing using the type II clustered regularly interspaced short palindromic repeats (CRISPR)-associated protein (Cas) system has been applied to edit the plant genome in several herbaceous plant species. However, it remains unknown whether this system can be used for genome editing in woody plants. In this study, we describe the genome editing and targeted gene mutation in a woody species, Populus tomentosa Carr. via the CRISPR/Cas9 system. Four guide RNAs (gRNAs) were designed to target with distinct poplar genomic sites of the phytoene desaturase gene 8 (PtoPDS) which are followed by the protospacer-adjacent motif (PAM). After Agrobacterium-mediated transformation, obvious albino phenotype was observed in transgenic poplar plants. By analyzing the RNA-guided genome-editing events, 30 out of 59 PCR clones were homozygous mutants, 2 out of 59 were heterozygous mutants and the mutation efficiency at these target sites was estimated to be 51.7%. Our data demonstrate that the Cas9/sgRNA system can be exploited to precisely edit genomic sequence and effectively create knockout mutations in woody plants.
原文链接:http://www.nature.com/srep/2015/150720/srep12217/pdf/srep12217.pdf

