邱丽娟团队成功克隆大豆耐盐基因
作物科学研究所大豆基因资源研究团队在大豆耐盐基因挖掘研究中取得重要进展。2014年12月12日,研究结果在《The Plant Journal 》杂志上以Featured Article发表题为“salinity tolerance in soybean is modulated by natural variation in GmSALT3”的封面文章。论文第一作者为关荣霞博士,邱丽娟博士和Matthew Gilliham博士为通讯作者。
土壤盐渍化是世界农业面临的一个重要问题,未来35年全球受盐渍化影响的土壤面积可能会加倍,而多数作物栽培品种属于盐敏感品种,在盐渍化条件下可导致作物大幅度减产。在耕地面积难以有效增加的情况下,通过培育耐盐品种应对环境胁迫是提高作物产量和解决粮食安全的有效途径。
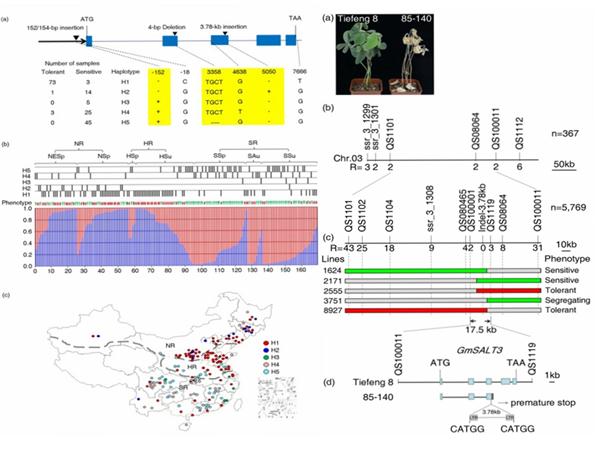
上世纪60年代美国科学家已经发现栽培大豆的耐盐性由一对显性基因控制。近十年美国、日本和韩国科学家陆续报道在栽培和野生大豆3号染色一个保守区间发现与大豆耐盐性相关的主效QTL,但一直未能图位克隆该基因。邱丽娟领导的大豆基因资源研究创新团队与澳大利亚阿德莱德大学科学家Matthew Gilliham研究小组合作,通过10多年的坚持不懈研究,建立了大豆耐盐性鉴定技术体系,开展了大豆耐盐基因资源鉴定和相关基因定位研究,最终通过图位克隆获得了栽培大豆铁丰8号的耐盐基因GmSALT3。该基因编码一个定位于内质网的离子转运蛋白。近等基因系的嫁接试验表明,该基因在根/下胚轴中控制钠离子在地上部的积累。盐敏感亲本85-140基因组中由于一个反转录转座子的插入导致基因功能的丧失。通过对微核心种质和野生大豆中该基因的编码序列分析,发现GmSALT3基因至少存在9种不同的单倍型(基因编码区变异类型),栽培大豆和野生大豆中分别有5种和8种不同的单倍型,其中只有铁丰8号类型(H1)和来自野生大豆的H7单倍型的为耐盐相关单倍型,其他7种为盐敏感相关单倍型。耐盐单倍型(H1)的分布与我国盐碱地的分布相吻合,并且在驯化过程中受到严格的选择。基因启动子区分析发现,H1和H7单倍型的序列高度相似,而除H2(敏感品种85-140类型)外的敏感单倍型在启动子区均有148bp或150bp的片段插入,H2类型第三外显子有3.8kb反转录转座子的插入,使得可以通过两个标记有效区分敏感单倍型与耐盐单倍型。
该研究明确了栽培和野生大豆中耐盐基因的变异类型,不仅为耐盐资源的标记辅助筛选提供了高效鉴别标记,也为进一步阐明大豆耐盐机理奠定了基础。
TPJ杂志为封面文章提供一个月免费下载:http://onlinelibrary.wiley.com/doi/10.1111/tpj.12695/abstract