【简化基因组产品家族大盘点】究竟谁才是最适合你的那个它?
经常看到简化基因组、RAD、GBS、dd-RAD、2b-RAD这些名词,但是具体指什么?相互之间的联系和区别是什么呢?是不是觉得有些糊涂呢?今天我就和大家捋一捋这些产品之间千丝万缕的关系。
简化基因组:通过限制性内切酶对基因组DNA进行酶切,并对酶切片段进行高通量测序,这种通过酶切降低基因组的复杂度的测序技术统称为简化基因组测序技术。下面这张图展现的是简化基因组这个大家族的成员:
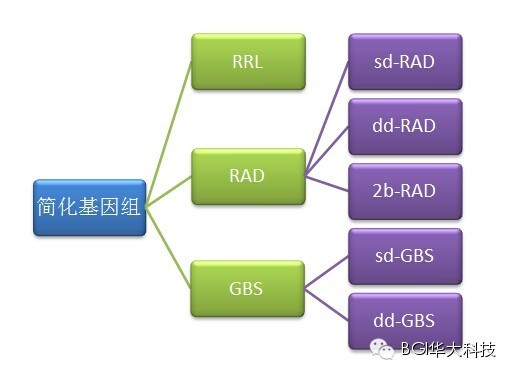
RRL(Reduced-RepresentationLibraries):简化文库,是最早的简化基因组技术,2000年由Altshuler, D.提出,最初应用于人类样品,后逐渐应用于动植物样品,由于其没有引入barcode ,不能区分混合样品,后续使用较少。RAD及GBS可以说是RRL的升级版。
RAD: (Restriction-site Associated DNA)是与限制性核酸内切酶识别位点相关的DNA。2007年由miller提出。
GBS:(Genotyping-by-Sequencing)是指通过测序进行基因分型。2011年由Elshire, R. J.提出。
虽然这几种技术原理比较相似,但是字面看不出联系,因为命名逻辑不同。后续虽然不同研究人员或测序机构在最初的方法上有改进甚至进行了重新命名,但是万变不离其宗,只要知道最本质的区别就能轻易将这些技术了解清楚。
下面的图片引自《Nat Rev Genet.》的权威综述:
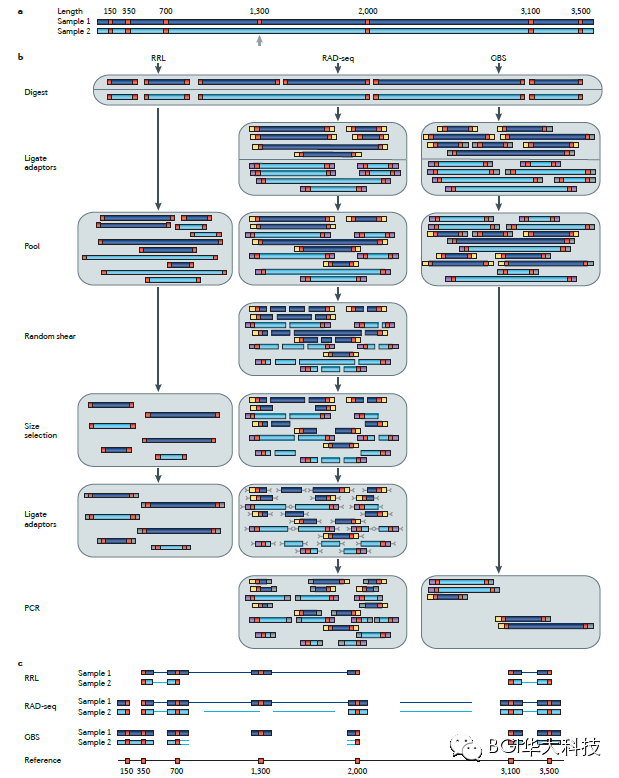
从上图的技术流程对比可以看出:
RRL的优点是步骤少,缺点是没有对不同样品加上不同的barcode,导致后续无法区分单个样品;
RAD-seq的技术优点是不同样品加上特有的barcode,且能把绝大部分酶切位点两侧的片段收集下来,缺点是增加了随机打断、片段选择、加Y型接头等步骤,稍微复杂些;
GBS 优点是不同样品加上的特有的barcode步骤简单,缺点是只能收集短的酶切片段,酶切片段偏少。
近几年简化基因组技术也在不断发展并逐渐分化出不同的分支,如RAD技术就在原来的sd-RAD(single digest-RAD,即常见的单酶切RAD)的基础上改进分化出dd-RAD、2b-RAD; GBS也在原来的sd-GBS(single digest-GBS,即单酶切GBS)的基础上分化出dd-GBS。
dd-RAD(doubledigest-RAD),即双酶切RAD,2012年由Brant K.提出。采用两种酶对基因组DNA进行酶切,第二种酶的酶切取代了随机打断的作用,优点是不同样品在同一酶切位点处收集的片段长度一致,缺点是获得的片段比单酶切RAD少。
2b-RAD(IIB-RAD),采用IIB型限制性内切酶进行酶切,来产生一致性片段进行测序,由王师教授2012年提出。IIB型限制性内切酶能在识别位点的上游和下游位点分别切段,获得固定长度的片段。优点是片段长度一致,缺点是片段长度较短,一般在33或36bp.
dd-GBS(doubledigest-GBS),即双酶切GBS,采用两种酶对基因组进行酶切,步骤与双酶切RAD非常相似,区别在于pooling的样品数量有所不同。
其实,每一种技术都有自己独特的应用领域,并不能简单的说优点和缺点,比如说GBS采用甲基化敏感酶时虽然收集的片段偏少,但是可以避开高重复区域,收集到的酶切片段及得到的标记有效性更高,比较适合高重复物种。双酶切RAD虽然获得的片段数量少,但是不同样品在同一酶切位点处收集的片段长度一致,可以将read2部分也利用起来,利于后续设计探针等,更适合无参考序列的物种。想知道哪一种方法更适合自己的项目需要结合物种情况及研究目的综合得出。
华大科技推出的几种常见简化基因组产品的特点如下:

大家赶快结合自己的课题特点及产品特色研究一下哪种产品更适合你吧。如果不清楚可以咨询我们,华大科技在简化基因组方面积累了深厚的项目经验,可以为您给出最专业的推荐!
其它如2b-RAD、dd-GBS等非常见项目也可以咨询我们。咨询电话:400-706-6615
参考文献:
1. Davey J W et al. Genome-wide genetic markerdiscovery and genotyping using next-generation sequencing. Nat Rev Genet.17,12(7):499-510(2011).
2. Altshuler, D. et al. An SNP map of the humangenome generated by reduced representation shotgun sequencing. Nature.407,513–516 (2000).
3. Miller, M. R. et al. Rapid andcost-effective polymorphism identification and genotyping using restrictionsite associated DNA (RAD) markers. Genome Res. 17, 240–248 (2007).
4. Elshire, R. J. et al. A robust, simplegenotyping by sequencing (GBS) approach for high diversity species. PLoS ONE 6,e19379 (2011).
5. Peterson BK et al. Double digest RADseq: aninexpensive method for de novo SNP discovery and genotyping in model andnon-model species. PLoS One. 7(5):e37135(2012).
6. WangS et al. 2b-RAD: a simple and flexible method for genome-wide genotyping. NatMethods. 20;9(8):808-10(2012).

