武汉植物园绘制首个水稻柱头组织磷酸化蛋白质图谱
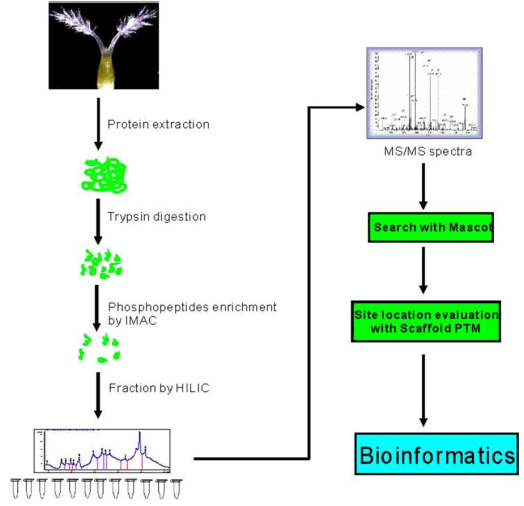
水稻柱头磷酸化蛋白质鉴定流程图
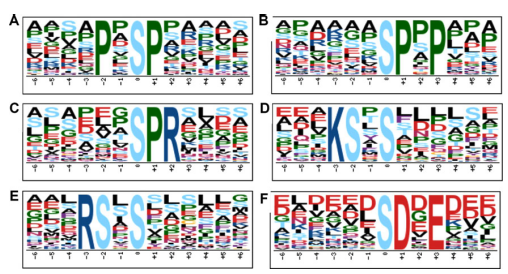
鉴定到的保守性磷酸化肽段序列
蛋白质作为生命体主要的生物功能执行者,其表达量的升高、降低、或者各种修饰都是伴随生物学过程的发生而不断变化着。蛋白质磷酸化修饰,是生物体内的一种普遍而重要的蛋白质翻译后修饰方式,通过在蛋白质的特定氨基酸上加入和去掉磷酸基团,从而改变蛋白质的构型、活性以及与其他分子的相互作用方式来调控生物体内多种生物学途径。近年来,植物组织的磷酸化蛋白质组学的研究越来越成为植物蛋白质组学的研究热点。
水稻作为禾本科种子植物,需要通过授粉产生种子进行下一代的繁衍。柱头作为水稻授粉过程的重要参与者之一,其组织所含蛋白质等各种物质参与到柱头授粉后花粉的识别及一系列的信号传递过程。中国科学院武汉植物园杨平仿研究员带领的资源植物繁殖生物学研究团队从事水稻的种子萌发及水稻柱头传粉受精相关工作,对水稻柱头蛋白质的磷酸化修饰进行深入研究,并取得相关研究成果。
近期该团队以王坤副研究员为第一作者,通过利用IMAC 磷酸化肽段富集技术对水稻成熟柱头组织蛋白质的磷酸化肽段进行富集,结合Triple TOF5600蛋白质质谱仪鉴定和多种生物信息学软件分析,共获得1588个柱头组织的磷酸化蛋白质,包括2347个磷酸化位点。其中包括654个水稻中新发现的磷酸化蛋白质,结合已有研究结果,共有201个磷酸化蛋白质属于柱头组织特异表达蛋白质。该研究结果不仅首次绘制了水稻柱头组织磷酸化蛋白质图谱,同时也将极大促进水稻柱头发育和授粉相关方面的研究。
该研究得到国家自然基金(31100230)和中国科学院“百人计划”经费,以及武汉大学开放课题经费(KF201403)共同支持。具体研究结果发表在Proteomics 期刊上。
原文摘要:
Analysis of phosphoproteome in rice pistil
Kun Wang, Yong Zhao, Ming Li, Feng Gao, Ming-kun Yang, Xin Wang, Shaoqing Li andPingfang Yang
As the female reproductive part of a flower, the pistil consists of the ovary, style, and stigma, and is a critical organ for the process from pollen recognition to fertilization and seed formation. Previous studies on pollen–pistil interaction mainly focused on gene expression changes with comparative transcriptomics or proteomics method. However, studies on protein PTMs are still lacking. Here we report a phosphoproteomic study on mature pistil of rice. Using IMAC enrichment, hydrophilic interaction chromatography fraction and high-accuracy MS instrument (TripleTOF 5600), 2347 of high-confidence (Ascore ≥ 19, p ≤ 0.01), phosphorylation sites corresponding to 1588 phosphoproteins were identified. Among them, 1369 phosphorylation sites within 654 phosphoproteins were newly identified; 41 serine phosphorylation motifs, which belong to three groups: proline-directed, basophilic, and acidic motifs were identified after analysis by motif-X. Two hundred and one genes whose phosphopeptides were identified here showed tissue-specific expression in pistil based on information mining of previous microarray data. All MS data have been deposited in the ProteomeXchange with identifier PXD000923 (http://proteomecentral.proteomexchange.org/dataset/PXD000923). This study will help us to understand pistil development and pollination on the posttranslational level.
作者:武汉植物园

