微生物所在PNAS揭示PILR受体识别唾液酸的分子机制
中科院微生物所首次系统的阐释了PILR受体识别唾液酸的分子机制,为了解PILR相关的配体结合铺平了道路。相关文章发表于2014年5月19日的《PNAS》杂志上。
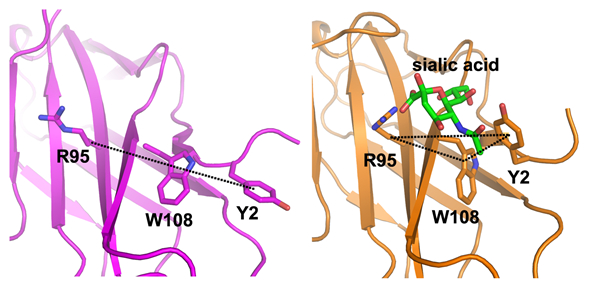
图注: PILR受体的“三残基”模块结合唾液酸后发生线性排列到三角排列的转变。
左图:唾液酸结合前;右图:唾液酸结合后。
成对免疫球蛋白样受体α和β(paired immunoglobulin-like type 2 receptor α/β, PILRα/PILRβ)是参与先天免疫调控的重要表面分子,在不同的物种中高度保守。两种分子均在胞外“暴露”相似的单个免疫球蛋白样结构域以结合相应的配体;但结合配体后,却利用不同的胞内结构域传递相反的免疫调节信号。PILRα通过自身的免疫受体酪氨酸抑制基序(ITIM)介导免疫抑制;而PILRβ可以结合含有免疫受体酪氨酸激活基序(ITAM)的接头蛋白DAP-12,从而实现免疫激活。正是由于两种受体分子传递重要的对立信号以调控宿主免疫反应,使其成为众多成对免疫受体中备受关注的“一对”。
小鼠PILRα和PILRβ天然配体是CD99;同时也有研究表明人单纯疱疹病毒通过表面糖蛋白gB 与PILRα相互作用侵入宿主细胞。但是PILRα和PILRβ对配体的结合均依赖于配体中的唾液酸分子。不同于典型的唾液酸结合蛋白,如唾液酸结合性免疫球蛋白样凝集素(siglec),PILR受体对唾液酸的亲合力很低,无法通过单个的唾液酸分子测定结合。这一特点限制了PILR与唾液酸的互作机制研究。另一方面,PILRα和PILRβ虽然在胞外结构域中的序列相似性很高,但有切实证据表明两者呈现出显著的唾液酸结合能力的不同,形成这种差异的分子机理仍然“悬而未决”。
为了探究PILR与唾液酸互作的分子基础,从而对上述科学问题给出精确的科学解释,高福研究团队通过结构生物学方法首先对PILRα和PILRβ的结构进行了深入研究。结果表明两种分子均具有典型的siglec-样折叠,但缺少siglec分子特有的二硫键,取而代之利用一组疏水互作稳定分子构象。
在PILR/唾液酸的互作研究中,研究人员建立了一种以I型单纯疱疹病毒(HSV-1)的gB蛋白为依托的表面等离子共振检测方法,并通过定点诱变,证实了PILRα分子表面的Y2,R95和W108所形成的“三残基”模块在唾液酸结合中的关键作用;而PILRβ正是由于该模块中的W108L突变导致其失去了与唾液酸互作的能力。通过进一步的复合物结构研究,高福研究团队还首次揭示了PILRα识别唾液酸的分子细节:与单体PILR结构相比,复合物结构中Y2氨基酸发生了显著的构象和空间位置的变化,从而使上述的“三残基”模块由唾液酸结合前的线性排列转变为唾液酸结合后的三角模式排列(见下图)。
这一研究首次系统的阐释了PILR受体识别唾液酸的分子机制,为了解PILR相关的配体结合铺平了道路。上述研究成果已发表在《美国科学院院报》上。
原文摘要:
PILRα and PILRβ have a siglec fold and provide the basis of binding to sialic acid
Qiong Lu, Guangwen Lu, Jianxun Qi,Han Wang, Yifang Xuan, Qihui Wang,Yan Li, Yanfang Zhang, Chunfu Zheng,Zheng Fan, Jinghua Yan,and george F. Gao
Paired immunoglobulin-like type 2 receptor α (PILRα) and β (PILRβ) belong to the PILR family and are related to innate immune regulation in various species. Despite their high sequence identity, PILRα and PILRβ are shown to have variant sialic acid (SA) binding avidities. To explore the molecular basis of this interaction, we solved the crystal structures of PILRα and PILRβ at resolutions of 1.6 Å and 2.2 Å, respectively. Both molecules adopt a typical siglec fold but use a hydrophobic bond to substitute the siglec-specific disulfide linkage for protein stabilization. We further used HSV-1 glycoprotein B (gB) as a representative molecule to study the PILR–SA interaction. Deploying site-directed mutagenesis, we demonstrated that three residues (Y2, R95, and W108) presented on the surface of PILRα form the SA binding site equivalent to those in siglecs but are arranged in a unique linear mode. PILRβ differs from PILRα in one of these three residues (L108), explaining its inability to engage gB. Mutation of L108 to tryptophan in PILRβ restored the gB-binding capacity. We further solved the structure of this PILRβ mutant complexed with SA, which reveals the atomic details mediating PILR/SA recognition. In comparison with the free PILR structures, amino acid Y2 oriented variantly in the complex structure, thereby disrupting the linear arrangement of PILR residues Y2, R95, and W108. In conclusion, our study provides significant implications for the PILR–SA interaction and paves the way for understanding PILR-related ligand binding.
作者:微生物所

