生物物理所在Cell子刊揭示羟甲基化DNA的特异识别机制
中国科学院生物物理研究所许瑞明研究组、朱冰研究组和华东师范大学翁杰敏研究组合作,揭示5-羟甲基化胞嘧啶(5hmC)被UHRF2-SRA结构域特异识别的分子机制,首次证实了UHRF2蛋白作为5hmC特异识别蛋白的存在(5hmC specific reader)。相关文章发表于2014年5月8日的《molecular Cell》杂志上。
生物物理所等揭示羟甲基化DNA的特异识别机制
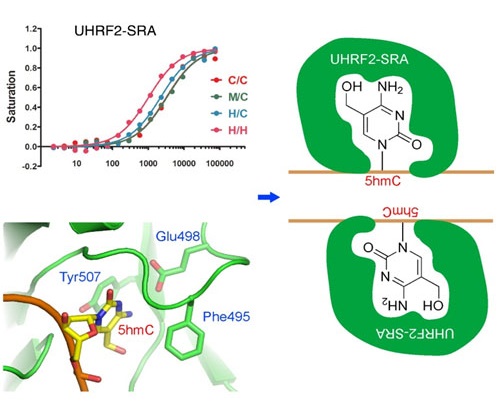
UHRF2的SRA结构域能特异识别并结合5hmC。从复合物的晶体结构中可以看出,SRA结合口袋中的苯丙氨酸(F495)对于5hmC的亲和性起着至关重要的作用,而NKP环则影响其对半甲基化与双边甲基化DNA的辨别。
5hmC是Tet蛋白介导的DNA主动去甲基化过程的第一步反应产物,丰度远高于其他两种中间产物(5fC、5caC)。高分辨率的基因组测序显示5hmC分布在基因调节区域,作为一种稳定的表观遗传学标记,在细胞分化、胚胎发育以及疾病发生过程中起着不可或缺的作用。2013年Cell杂志报道了UHRF2的SRA结构域可以特异识别5hmC,但具体的识别机理并不清楚。
此项研究中,研究团队解析了UHRF2-SRA与含有5hmC、5mC和C的多种DNA复合物的晶体结构。分析不同修饰的胞嘧啶在SRA结合口袋中的差异,研究团队揭示了5hmC的羟基与Thr508形成的一个多余氢键,是UHRF2-SRA对5hmC具有更强亲和力的原因。比较UHRF2-SRA-5hmC与UHRF1-SRA-5mC的结构,研究团队发现两个氨基酸的差异(F495E498对应Y471D474)是UHRF2-SRA特异识别5hmC的结构基础。另外一个有趣的发现是,相对于UHRF1-SRA特异识别半甲基化的DNA(hemi-modified),UHRF2-SRA更偏好结合双边修饰的DNA(fully-modified),研究团队进一步通过一种双边碱基都翻转出来的复合物结构阐明了UHRF2 的NKR loop区域对其双边修饰选择性的影响。这些结果对于更加深入地理解5hmC的功能起着重要的推动作用。
该研究得到了科技部“973”计划、基金委重大研究计划以及中科院战略性先导科技专项(B类)等的资助。
原文摘要:
Structural Basis for Hydroxymethylcytosine Recognition by the SRA Domain of UHRF2
Ting Zhou, Jun Xiong, Mingzhu Wang, Na Yang, Jiemin Wong, Bing Zhu, Rui-Ming Xu
Methylated cytosine of CpG dinucleotides in vertebrates may be oxidized by Tet proteins, a process that can lead to DNA demethylation. The predominant oxidation product, 5-hydroxymethylcytosine (5hmC), has been implicated in embryogenesis, cell differentiation, and human diseases. Recently, the SRA domain of UHRF2 (UHRF2-SRA) has been reported to specifically recognize 5hmC, but how UHRF2 recognizes this modification is unclear. Here we report the structure of UHRF2-SRA in complex with a 5hmC-containing DNA. The structure reveals that the conformation of a phenylalanine allows the formation of an optimal 5hmC binding pocket, and a hydrogen bond between the hydroxyl group of 5hmC and UHRF2-SRA is critical for their preferential binding. Further structural and biochemical analyses unveiled the role of SRA domains as a versatile reader of modified DNA, and the knowledge should facilitate further understanding of the biological function of UHRF2 and the comprehension of DNA hydroxymethylation in general.
作者:中科院

