DNA测序和宏基因组学助力颗粒物研究
发表日期:2014-02-15 08:30PM 阅览次数:
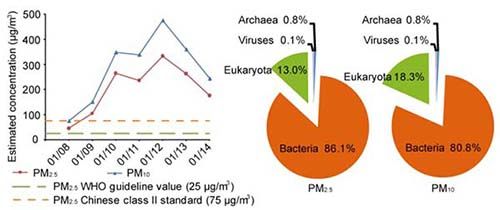
图为2013年1月8日~14日测得的PM2.5日平均浓度(左),以及测序分析得到的DNA序列的生物域分布(右)。
1月23日,清华大学生命学院朱听研究组在环境学杂志 Environmental Science & Technology在线发表了题为Inhalable microorganisms in Beijing’s PM2.5 and PM10 pollutants during a severe smog event的研究论文。该论文报道了北京市雾霾天气中大气悬浮颗粒物的微生物组分。
大气悬浮颗粒物作为影响我国大多数城市空气质量的首要污染物,已逐渐成为全社会关心的影响公共健康的重大问题。PM2.5(空气动力学直径≤2.5 µm的细颗粒物)和PM10颗粒物(空气动力学直径≤10 µm的粗颗粒物)悬浮在空气中,成为诸多微生物、有毒有害化学物质侵入人类呼吸道的载体。虽然目前DNA测序和宏基因组学方法已经被广泛用于环境微生物研究,但将其应用于研究大气悬浮颗粒物的微生物组分,一直存在颗粒物样品DNA含量少、用传统方法研究难以获得足够的DNA进行测序等困难。朱听研究组在研究中建立了一套从大气悬浮颗粒物样品中提取、纯化DNA,并进行测序与宏基因组学分析的技术。该研究首次在“种”的水平上鉴别出大气悬浮颗粒物中的微生物,发现其中大部分为非致病性微生物,并且有很多可能来自于土壤。该结果有望为相关的公共医学、城市规划和雾霾治理等研究提供有用的数据。
该研究与清华大学环境学院蒋靖坤研究组、清华大学测序平台田埂研究组合作完成。

