薛定研究组在Cell Research报道凋亡DNA降解通路研究新成果
2013年12月10日,清华大学生命科学学院薛定教授研究组在《自然》子刊Cell Research上发表学术论文“A novel mechanism underlies caspase-dependent conversion of the dicer ribonuclease into a deoxyribonuclease during apoptosis”,通过生化、功能与结构模拟分析揭示了秀丽线虫中RNase III家族成员DCR-1蛋白(dicer)在细胞凋亡过程中转化为DNA酶的机制。薛定教授为该论文通讯作者,生命科学学院博士生葛晓、清华-北大-NIBS三校联合项目研究生赵翔、美国科罗拉多大学Akihisa Nakagawa博士为本研究论文的共同第一作者。
RNase III家族是一类双链RNA特异性的核酸酶,其中dicer蛋白是miRNA生成以及RNAi通路上的重要环节。2010年,薛定教授研究组发现秀丽线虫的dicer蛋白DCR-1可在凋亡蛋白酶CED-3的作用下转化为DNA酶,启动染色体DNA的降解,促进细胞凋亡。这一研究作为封面文章发表在当年的Science期刊上(Science 2010: Vol. 328, 327-334)。
在线虫细胞凋亡过程中,CED-3可切割DCR-1,产生的羧基端截短产物tDCR-1具有DNA酶的活性,可使DNA产生带3’-羟基的缺口,这些缺口在下游DNA酶如CPS-6、CRN-1等的作用下使得染色体DNA进一步被降解;功能分析也表明tDCR-1在线虫体内具有DNA酶活性,并能恢复dcr-1基因缺失造成的凋亡缺陷表型。 但是,tDCR-1的这种DNA酶活性在全长DCR-1蛋白中是如何被抑制的,以及DCR-1由RNA酶向DNA酶转变的机制并不清楚。
该研究通过生化实验和体内功能检测发现,在DCR-1全长蛋白中,位于CED-3酶切位点之前的氨基酸序列并非全部为抑制tDCR-1的DNA酶活性所必需。当这些氨基端序列截短到一定程度时,就失去了对DNA酶活性的抑制,而这一抑制很可能是通过阻碍截短蛋白与DNA的结合实现的。 该研究还发现,tDCR-1的DNA酶活性也可被具有刚性二级结构的位于tDCR-1氨基端的无关蛋白序列抑制,揭示了tDCR-1基于空间构象,而非序列特异性的抑制机制。结构模拟也表明,CED-3酶切之后,DCR-1构象发生了显著变化,形成了适宜结合DNA的沟槽,而能抑制DNA酶活性的关键氨基端序列则位于阻碍构象变化的位置。该研究还预测RNase III家族的其他成员可通过与帮助结合DNA的结构单元连接的方式获得DNA酶活性,并通过构建tDCR-1/RNaseIII融合蛋白的方式进行了验证。
该研究阐释了DCR-1蛋白在细胞凋亡过程中由RNA酶向DNA酶转变的机制,并提示其他以RNA为底物的酶或者RNA结合蛋白,也可能通过类似机制调节RNA/DNA的底物特异性,参与更广泛的生化调控活动,促进显著的生理变化。
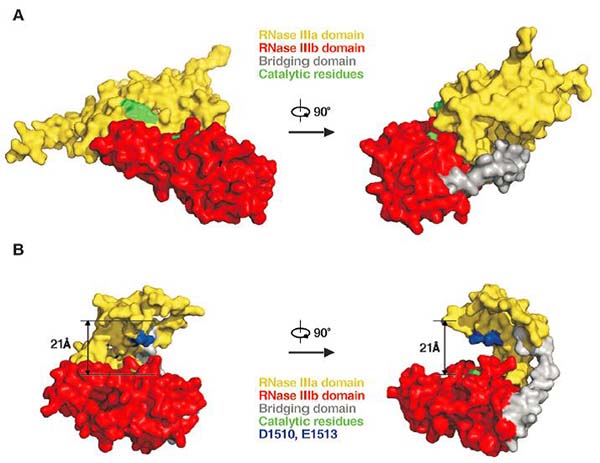
图示为DCR-1蛋白在CED-3酶切后构象变化的模型图
该成果与清华大学生命科学学院王新泉研究组和龚海鹏研究组合作完成,并利用筹建中的清华大学信息科学与技术国家实验室高性能计算平台开展了数据运算工作。

