Nature:活细胞DNA结构依赖ANP32E蛋白维持
新加坡国立癌症中心、法国国家科研中心等处的科学家在转基因小鼠研究中,证实活细胞中的DNA结构在一定程度上是通过一种叫做ANP32E的蛋白进行维持。ANP32E是一种人类蛋白质。相关文章发表于2014年1月22日的《Nature》杂志上。
Nature:活细胞DNA结构依赖ANP32E蛋白维持
新加坡国立癌症中心炎症生物学实验室Patrick Reilly博士说:“尽管DNA通常被描述为双链螺旋结构,细胞中实际包含的DNA总量意味着这些DNA链必须被压缩成称之为染色质的复杂结构,在限制DNA体积的同时确保能够读取到编码信息。在大量的发育疾病和各种类型的癌症中都发现存在染色质结构错误。通过了解控制染色质结构的这些过程,我们希望能够改良我们的工具,以实现实时调控特异的基因,由此逆转许多疾病所造成的影响,开发出新型抗癌药物。”
在过去的数十年里,科学家们发现适当的基因调控在一定程度上依赖于这些结构。以往的研究证实一种叫做H2A.Z的特殊染色质元件在控制特异区域的DNA表达中发挥重要作用。H2A.Z是在所有真核生物中高度保守的一种组蛋白变体,参与保护常染色体、防止形成异染色质;并且与转录调节、抗沉默、沉默和基因组稳定有关。组蛋白变体H2A.Z可能与染色体形成独立的结构域,从而调节染色质结构功能。
酸性和磷酸蛋白32(ANP32家族)是含有亮氨酸重复结构(LRR)的蛋白质超家族。其参与了广泛的生物学过程:激素受体的相互作用,酶的抑制作用,细胞的粘附、转运、基因表达的调控,哺乳动物的早期发育、神经系统的发育、凋亡信号的转导、细胞骨架形态和动态的调控。近年来的研究发现ANP32家族成员ANP32E除了具有家族成员的保守功能外,还参与了细胞的增殖和肿瘤的发生与转移。多年来Reilly博士在新加坡国立癌症中心一直从事ANP32E蛋白研究。
在这篇新文章中,法国的研究人员发现ANP32E能够将DNA上的H2A.Z除去,从而改变基因表达。在研究合作中,Reilly博士提供了一种特异性缺失ANP32E的小鼠,研究人员发现缺失ANP32E的细胞丧失了正确的染色质结构。长期以来,Reilly博士都是基于ANP32E不同寻常的蛋白质成分,将它视作为一种可能的治疗靶点进行研究。从长远来看,这一研究发现有可能能够揭示出一些治疗新策略。
原文摘要:
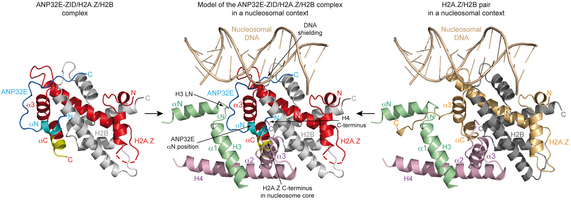
ANP32E is a histone chaperone that removes H2A.Z from chromatin
Arnaud Obri, Khalid Ouararhni, Christophe Papin, Marie-Laure Diebold, Kiran Padmanabhan, Martin Marek, Isabelle Stoll, Ludovic Roy, Patrick T. Reilly, Tak W. Mak,Stefan Dimitrov, Christophe Romier & Ali Hamiche
H2A.Z is an essential histone variant implicated in the regulation of key nuclear events. However, the metazoan chaperones responsible for H2A.Z deposition and its removal from chromatin remain unknown. Here we report the identification and characterization of the human protein ANP32E as a specific H2A.Z chaperone. We show that ANP32E is a member of the presumed H2A.Z histone-exchange complex p400/TIP60. ANP32E interacts with a short region of the docking domain of H2A.Z through a new motif termed H2A.Z interacting domain (ZID). The 1.48 Å resolution crystal structure of the complex formed between the ANP32E-ZID and the H2A.Z/H2B dimer and biochemical data support an underlying molecular mechanism for H2A.Z/H2B eviction from the nucleosome and its stabilization by ANP32E through a specific extension of the H2A.Z carboxy-terminal α-helix. Finally, analysis of H2A.Z localization in ANP32E−/− cells by chromatin immunoprecipitation followed by sequencing shows genome-wide enrichment, redistribution and accumulation of H2A.Z at specific chromatin control regions, in particular at enhancers and insulators.

