我校揭示DNA腺嘌呤去甲基化酶参与PRC2介导的基因表达抑制的新机制
南湖新闻网讯 (通讯员 张昕冉) DNA 腺嘌呤第六位氮原子上的甲基化(6mA)修饰最早发现广泛存在于原核生物基因组中,保护基因组免受外源DNA入侵。近年来,多种真核生物基因组DNA上的6mA 修饰也被发现,且其在基因组中的分布、丰度和功能存在显著差异。由于不同物种的基因组、染色质结构以及染色质修饰方式的不同,6mA可作为转录激活或抑制的表观标记发挥作用,可以参与干细胞发育、非生物胁迫的响应等。6mA修饰一般由腺嘌呤甲基转移酶METTL和去甲基化酶ALKBH进行动态调控。DNA腺嘌呤去甲基化酶ALKBH1蛋白属于Fe(II)-α-酮戊二酸双加氧酶家族中AlkB亚家族成员,可以从单链或未配对的DNA中去除6mA。
多梳蛋白复合体PRC2催化的组蛋白H3第27位赖氨酸三甲基化(H3K27me3)是一类维持基因和基因组沉默的关键组蛋白修饰,在动植物生长发育和环境适应性应答中发挥着重要的调控作用。在水稻中,大约有8%的基因上存在H3K27me3和H3K4me3共定位的二价修饰,这种二价修饰使发育及胁迫响应相关的调节基因处于“蓄势待发”的瞬时激活状态,以便快速响应外界的信号变化。但是该二价修饰的动态变化是否会受到DNA腺嘌呤去甲基化酶的调控还从未被探究。
8月30日,华中农业大学作物遗传改良全国重点实验室、湖北洪山实验室水稻团队周道绣教授课题组在国际期刊Genome Biology上在线发表了题为“A DNA adenine demethylase impairs PRC2-mediated repression of genes marked by a specific chromatin signature”的研究论文,揭示了水稻DNA腺嘌呤去甲基化酶ALKBH1通过调节组蛋白H3K27me3/H3K4me3修饰之间的平衡从而控制基因表达的功能,为学界理清PRC2复合物介导H3K27me3调控基因表达的机制提供了新见解。
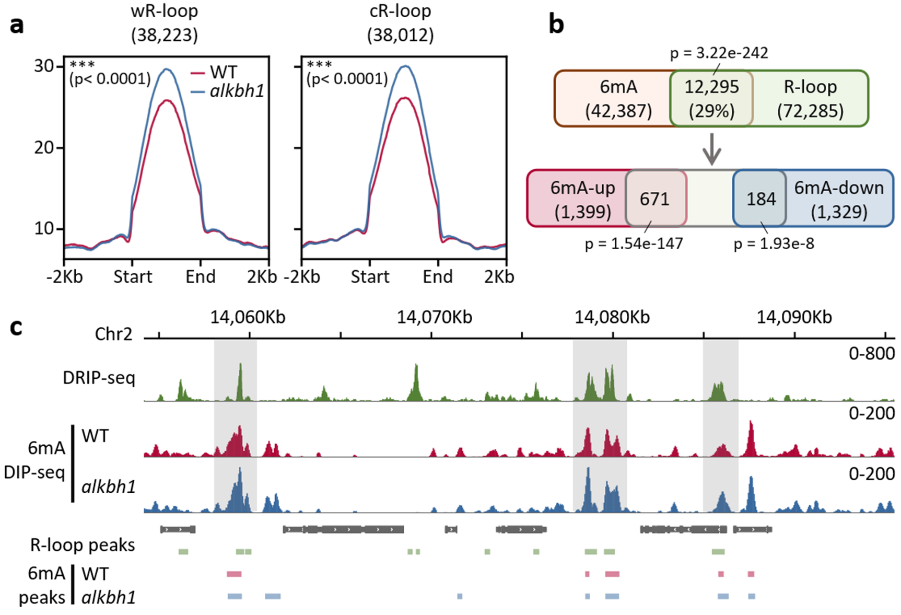
图1. ALKBH1调控水稻全基因组中R-loop上的6mA
该课题组前期研究首次揭示了DNA腺嘌呤甲基化修饰在水稻基因组中的分布特征与潜在功能,并证实了ALKBH1蛋白具有DNA腺嘌呤去甲基化酶活性,主要对单链DNA上的腺嘌呤进行去甲基化,并解析了ALKBH1的蛋白晶体结构。为了进一步探究ALKBH1在植物染色质中的功能,该团队首先对alkbh1突变体和野生型(WT)水稻14天幼苗材料进行了6mA全基因组免疫共沉淀测序(6mA-IP-seq),发现ALKBH1缺失突变对水稻全基因组6mA的影响较小,结合R-loop数据分析发现ALKBH1突变后R-loop上的6mA修饰显著上升,说明ALKBH1在水稻基因组中主要去除R-loop上的6mA(图1)。
随后对这两种材料进行了全基因组转录组测序(RNA-seq)和ALKBH1自身抗体的染色质免疫共沉淀测序(ChIP-seq),结合已发表的相同时期材料的表观修饰数据进行分析,发现ALKBH1激活具有特定染色质状态(H3K4me3与H3K27me3共定位且CG甲基化低)基因表达的功能,并且这些基因在胁迫响应方面显著富集。因此推测ALKBH1可能通过调节基因上H3K27me3/H3K4me3的比例从而调控基因表达。为了验证这一猜想,通过检测alkbh1突变体和野生型材料中H3K27me3,H3K4me3和H3K9me2的修饰水平发现ALKBH1突变后只有H3K27me3修饰水平显著上升,H3K4me3和H3K9me2修饰水平无明显变化。这两种材料H3K27me3 ChIP-seq与本组之前PRC2复合物核心成分EMF2b的ChIP-seq数据关联分析,发现ALKBH1与EMF2b在基因组中存在61%的共定位,并且当ALKBH1敲除后这些基因上的H3K27me3的修饰水平显著上升。结合ChIP-qPCR进一步证明了ALKBH1在基因组上可以拮抗EMF2b的沉积,使靶基因保持较高的H3K27me3/H3K4me3比例,从而维持基因的表达。
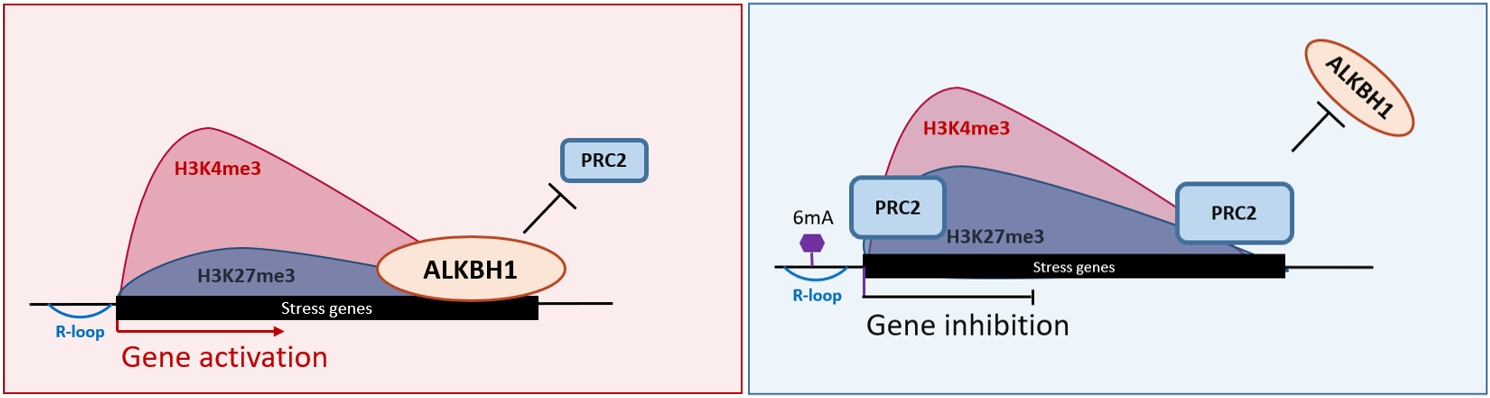
图2 ALKBH1拮抗PRC2调控胁迫响应基因的分子机制
综上所述,本研究发现进化上保守的ALKBH1在水稻基因组中可以去除R-loop上的6mA,并通过拮抗PRC2与目标基因的结合抑制H3K27me3,降低H3K27me3/H3K4me3比率,进而促进基因表达。这些结果揭示了ALKBH1 的新功能,并为 PRC2 控制基因表达的调控机制提供了新的见解。
华中农业大学作物遗传改良全国重点实验室、湖北省洪山实验室和生命科学技术学院博士研究生贾庆霄和张昕冉为论文共同第一作者,周道绣教授为文章通讯作者,袁猛教授和赵毓教授为本研究的工作提供了指导和帮助。王文韬博士,马瑄博士,李胜硕士,博士研究生刘潜、李俊杰、朱波、田晶晶以及硕士研究生龚世诚也参与了该研究工作。该研究得到国家自然科学基金项目的支持以及中央高校基本科研业务费等项目的资助。
【英文摘要】
The Fe (II)- and α-ketoglutarate‑dependent AlkB family dioxygenases are implicated in nucleotide demethylation. AlkB homolog1 (ALKBH1) is shown to demethylate DNA adenine methylation (6mA) preferentially from single-stranded or unpaired DNA, while its demethylase activity and function in the chromatin context are unclear. Here, we find that loss-of-function of the rice ALKBH1 gene leads to increased 6mA in the R-loop regions of the genome but has a limited effect on the overall 6mA level. However, in the context of mixed tissues, rather than on individual loci, the ALKBH1 mutation or overexpression mainly affects the expression of genes with a specific combination of chromatin modifications in the body region marked with H3K4me3 and H3K27me3 but depleted of DNA CG methylation. In the similar context of mixed tissues, further analysis reveals that the ALKBH1 protein preferentially binds to genes marked by the chromatin signature and has a function to maintain a high H3K4me3/H3K27me3 ratio by impairing the binding of Polycomb repressive complex 2 (PRC2) to the targets, which is required for both the basal and stress-induced expression of the genes. Our findings unravel a function of ALKBH1 to control the balance between the antagonistic histone methylations for gene activity and provide insight into the regulatory mechanism of PRC2-mediated H3K27me3 deposition within the gene body region.
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-023-03042-4
审核人:赵毓

