我校在鲜切花采后衰老的转录后水平调控分子机制研究中取得新进展
南湖新闻网讯(通讯员 孙政)近日,我校果蔬园艺作物种质创新与利用全国重点实验室和湖北洪山实验室张帆教授课题组揭示了转录后水平调控康乃馨鲜切花采后衰老的分子机制,研究以“An insertion of transposon in DcNAP inverted its function in the ethylene pathway to delay petal senescence in carnation (Dianthus caryophyllus L.)”为题发表在Plant Biotechnology Journal上。
花卉是极其重要的园艺作物,在满足人们的精神需求、服务乡村振兴和美丽中国建设、巩固拓展脱贫攻坚成果中发挥着越来越关键的作用。康乃馨(Carnation)学名为香石竹(Dianthus caryophyllus L.),是世界四大鲜切花之一,也是我国最重要的鲜切花之一,具有极高的观赏价值和经济价值。康乃馨同时也是一种典型的乙烯敏感性切花,被认为是研究乙烯调控鲜切花采后衰老,特别是花瓣衰老的模式植物。课题组的前期研究系统阐明了乙烯在转录水平和翻译后水平调控康乃馨花瓣衰老的分子机制,近期研究也揭示了表观遗传修饰调控康乃馨花瓣衰老的分子机理,但对于乙烯在转录后水平如何调控康乃馨花瓣衰老方面的知识仍有待研究。
为分析乙烯在转录后水平如何调控康乃馨花瓣衰老,本研究对课题组前期构建的乙烯处理不同时间的康乃馨花瓣衰老进程转录组数据进行分析,发现NAC家族转录因子DcNAP在康乃馨花瓣衰老过程中能够快速响应乙烯并显著诱导表达。通过进一步的研究,团队发现康乃馨基因组上存在一对DcNAP等位基因(DcNAP,DcNAP-dTdic1),其中DcNAP-dTdic1等位基因的第二个外显子上插入了一个886bp大小的dTdic1转座子。DcNAP等位基因能够编码含有完整NAC结构域的DcNAP1蛋白,而dTdic1转座子的插入则破坏了DcNAP-dTdic1等位基因的结构,并通过选择性剪接产生NAC结构域发生缺失的DcNAP2蛋白。
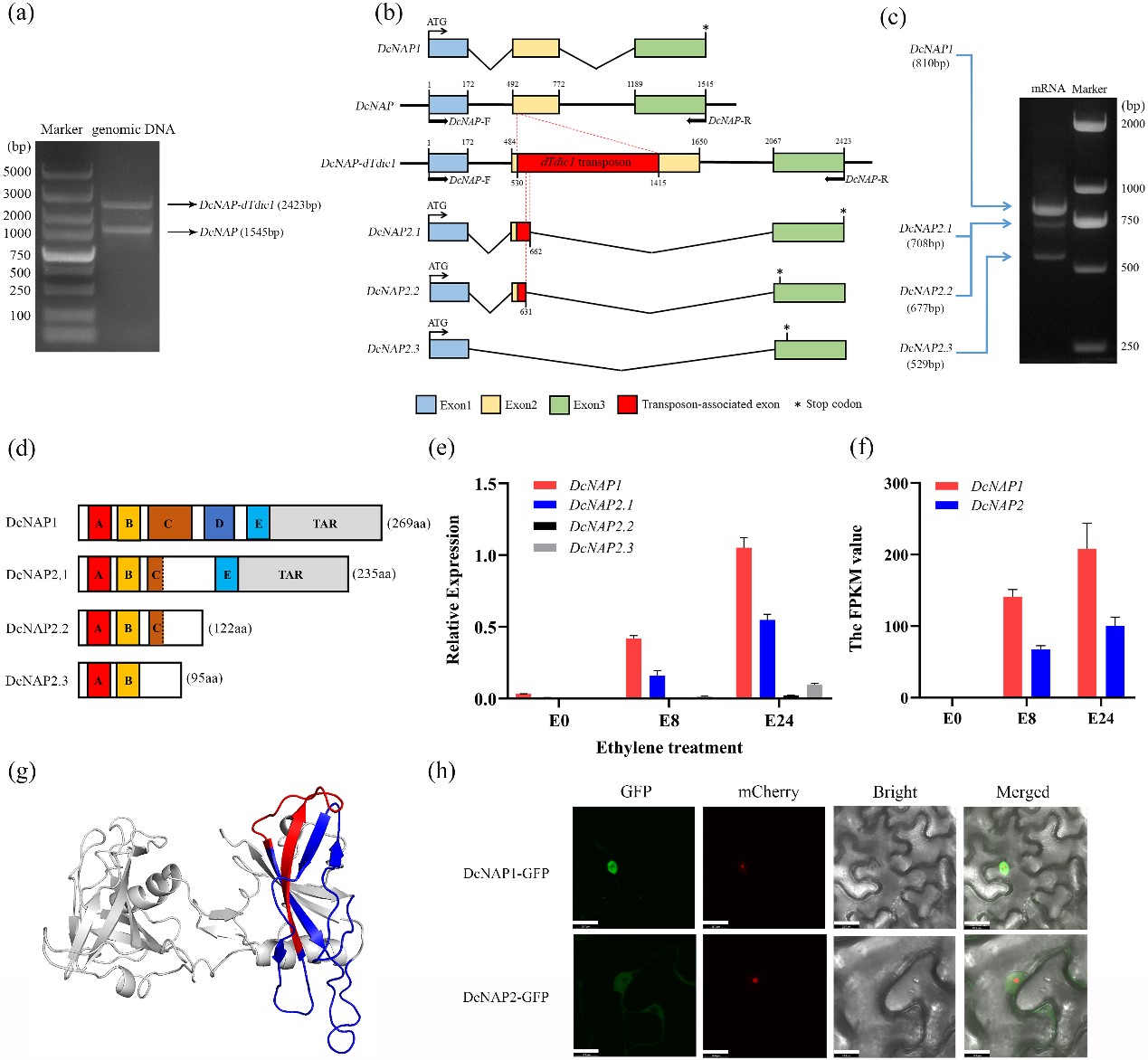
图1 dTdic1转座子插入导致衰老关键转录因子DcNAP发生结构变异
研究发现,通过VIGS技术瞬时沉默DcNAP1能够明显延缓康乃馨花瓣的衰老进程,同时乙烯生物合成关键基因和衰老相关基因的表达量显著下降;而瞬时沉默DcNAP2能够明显加速康乃馨花瓣的衰老进程,同时乙烯生物合成关键基因和衰老相关基因的表达量显著上升。这表明DcNAP1和DcNAP2在调控康乃馨花瓣衰老的过程中行使着相反的功能。
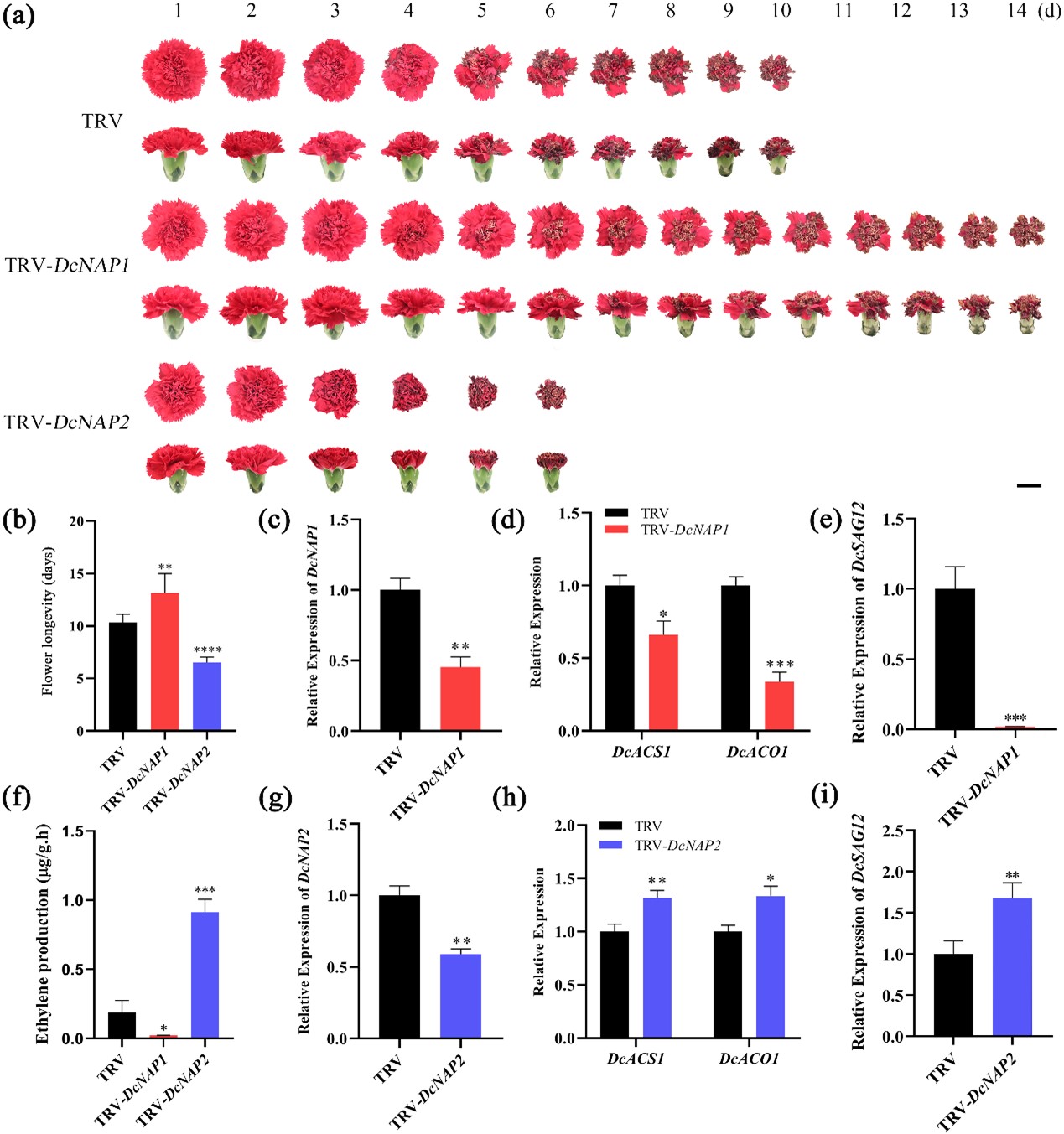
图2 沉默DcNAP1和DcNAP2能够分别延缓和加速康乃馨花瓣的衰老进程
进一步的研究发现,DcNAP1能够结合到乙烯生物合成关键基因的启动子上并激活它们的表达, 而DcNAP2则不能结合到乙烯生物合成关键基因的启动子上;但DcNAP2能够与DcNAP1发生直接的相互作用,从而拮抗DcNAP1对乙烯生物合成关键基因的激活作用。同时,团队发现,乙烯信号通路核心转录因子DcEIL3-1能够同时结合到DcNAP1和DcNAP2的启动子上并激活它们的表达。此外,DcNAP-dTdic1等位基因在不同乙烯敏感性康乃馨品种间存在差异,暗示DcNAP-dTdic1等位基因可以作为开发筛选不同乙烯敏感性康乃馨品种的分子标记。

图3 DcNAP2拮抗DcNAP1对乙烯生物合成关键基因的激活作用
团队通过研究阐明了乙烯在转录后水平调控康乃馨花瓣衰老的分子机制,能够进一步拓宽人们对康乃馨花瓣衰老分子调控网络的认识。这为筛选和培育货架期及瓶插期延长的康乃馨新品种奠定了关键的理论基础,为分子标记开发提供重要的基因资源。同时,本研究为人工操纵NAC转录因子的NAC结构域进行作物遗传改良提供了有效策略。
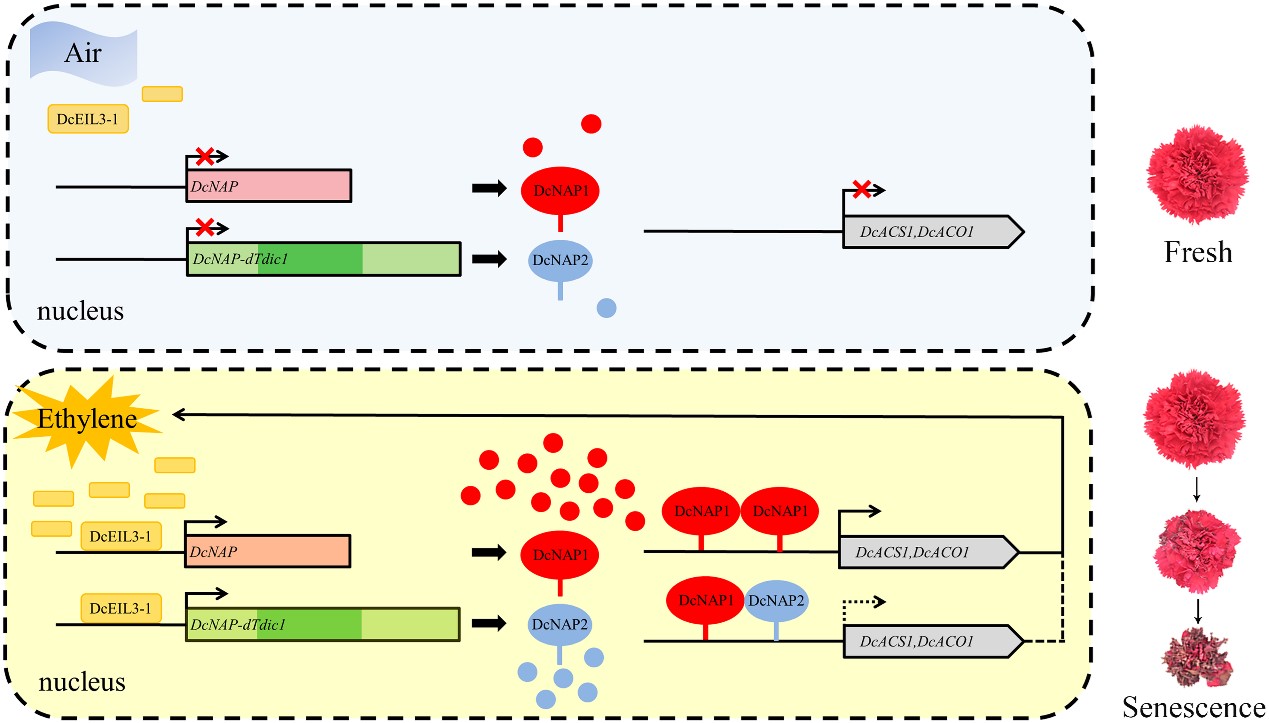
图4 乙烯在转录后水平调控康乃馨花瓣衰老的工作模型
我校果蔬园艺作物种质创新与利用全国重点实验室博士研究生孙政为论文第一作者,张帆教授为论文通讯作者,程运江教授和包满珠教授参与了本象研究。研究得到中央高校基本科研业务费专项基金(2662019PY049)、华中农业大学高层次人才启动经费项目和华中农业大学中央高校优秀青年团队培育项目(2662023PY011)等的资助。
【英文摘要】
Petal senescence is the final stage of flower development. Transcriptional regulation plays key roles in this process. However, whether and how post-transcriptional regulation involved is still largely unknown. Here, we identified an ethylene-induced NAC family transcription factor DcNAP in carnation (Dianthus caryophyllus L.). One allele, DcNAP-dTdic1, has an insertion of a dTdic1 transposon in its second exon. The dTdic1 transposon disrupts the structure of DcNAP and causes alternative splicing, which transcribes multiple domain-deleted variants (DcNAP2 and others). Conversely, the wild type allele DcNAP transcribes DcNAP1 encoding an intact NAC domain. Silencing DcNAP1 delays and overexpressing DcNAP1 accelerates petal senescence in carnation, while silencing and overexpressing DcNAP2 have the opposite effects, respectively. Further, DcNAP2 could interact with DcNAP1 and interfere the binding and activation activity of DcNAP1 to the promoters of its downstream target ethylene biosynthesis genes DcACS1 and DcACO1. Lastly, ethylene signalling core transcriptional factor DcEIL3-1 can activate the expression of DcNAP1 and DcNAP2 in the same way by binding their promoters. In summary, we discovered a novel mechanism by which DcNAP regulates carnation petal senescence at the post-transcriptional level. It may also provide a useful strategy to manipulate the NAC domains of NAC transcription factors for crop genetic improvement.
论文链接:https://onlinelibrary.wiley.com/doi/10.1111/pbi.14132
审核人:张帆

