由结构域边界插入来调节基因组的空间折叠
北京时间2020年8月31日晚23时,美国费城儿童医院和宾夕法尼亚大学医学院的Gerd Blobel教授,其实验室的张帝(Di Zhang),和他们的合作者在Nature Genetics上发表了题为Alteration of genome folding via contact domain boundary insertion的论文,报道了通过运用一系列基因组编辑技术来改变基因组三维折叠,构建新的亚微米结构,并剖析边界DNA元件和不同基因组环境间相互作用的发现。
哺乳动物基因组DNA的线性距离可长达~2米,而包含其的细胞核仅有几微米宽。之所以DNA可被压缩106(兆)倍进而储藏于细胞核中,是因为基因组在三维空间中经过错综复杂的折叠和组织。
三维基因组学(3D genomics/genome architecture)研究的焦点之一是在亚微米(sub-micrometer)及其对应的兆碱基(megabase)/亚兆碱基(sub-megabase)量级上结构和功能间的联系。
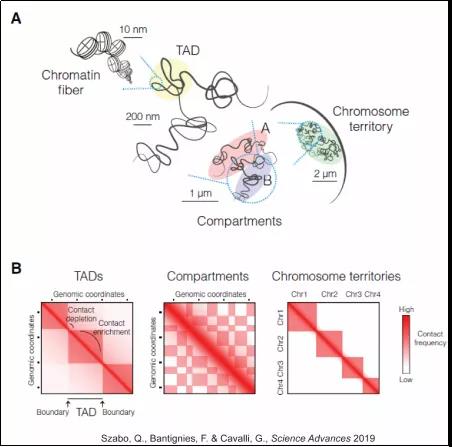
这些结构是由高通量染色质构象捕获技术(ahigh-throughput, genome-wide version of chromosome conformation capture: Hi-C)描述的,可大致分为(如图):
1. 染色质区室化 (compartmentalization):传统上被认为是兆碱基量级上开放的(转录活跃)染色质(open chromatin: A compartment)和封闭的(转录不活跃)染色质(closed chromatin: B compartment)。属于同种区室的染色质区域相互接触,而异种区室间相互隔开。因而在Hi-C热图(heatmap)上显现格子状(plaid)的图案。
2. 拓扑结构域(topologically associated domain, TAD)或接触结构域(contact domain): 兆碱基-亚兆碱基级别,结构域内的染色质倾向于相互接触,而属于两个不同结构域染色质之间的跨边界(domain boundary)接触相对稀少。Hi-C热图上对应沿对角线上的三角/正方形。
异常的结构域组织可涉及发育障碍,肢体畸形和癌症等疾病。精确的结构域组织一定程度上取决于架构因子(architectural factors),其中被详尽研究的是一个多功能转录因子:CCCTC结合因子(CCCTC binding factor, CTCF)和黏连蛋白或粘合素(cohesin)。
事实上,一个主要解释染色质结构域形成的理论:环挤压模型(loop extrusion model)认为粘合素沿着染色质进行挤压,形成逐渐增大的环状结构,待其遇到一对呈会聚去向(convergently oriented)的CTCF而停滞。这一对CTCF和cohesin也因而一起划定结构域的边界(domain boundary)。
但值得一提的是,不是所有CTCF都处于结构域的边界,而且同样处于结构域边界的还有不少其他基因组特征(genomic features),比如转录起始位点(transcription start site: TSS)。目前不少描述性研究把一些基因组特征与其处于相同位置的结构域边界大致关联(correlation)起来。
然而,一些重要的因果机制(causation)问题还是没有得到彻底的解答。到底哪些DNA元件是形成结构域边界的必要和充分(对于后者的研究更少)条件?如果一个DNA元件在其原本的基因组位点上可以划定结构域边界,当它被放置于另一个基因组环境(genomic context)时,是否还能形成结构域边界?形成结构域边界的功能是怎么编码入DNA元件的,而有多大程度又被该元件周围的基因组环境所调节?
在一个近乎单倍体的人类细胞系中,张帝和他们的合作者先运用人工转座子(artificial transposon)把一段2千碱基位于结构域边界的DNA--其包含一个CTCF结合位点(CTCF binding site: CBS)和一个转录起始位点(TSS)--随机插入11个染色体共16个基因组位点。接着通过Hi-C来同时测量这同一个DNA元件怎样在不同基因组环境下影响染色质的空间架构。随后,作者更深入地用CRISPR解析了CTCF结合位点和转录起始位点各自以及共同地对于基因组折叠的功效。
文章主要结论
1. 通过筛选具有多个基因组位点被同时编辑的单细胞克隆,可实现一轮基因组编辑结合Hi-C对多个编辑位点的多重架构测定。
2. 该CBS-TSS元件在至少4个不同位点形成了新的接触结构域,造成了染色质区室转换,划分了新的边界,或者形成了新的反映环挤压的Hi-C表型(结构条纹:architectural stripes)。这个发现证明一个2千碱基(kb)的DNA元件可以用来改变其周围千兆碱基(10 Mbp)量级染色质的空间架构。
3. 该元件当插入本已存在的结构域边界时,可进一步加强边界。
4. 该元件在若干个位点没有导致明显的结构域水平上的变化,但深入探究后发现该元件还是造成了结构域内部的细微调整。巧合的是这些位点本已经有着相当复杂的架构格局。
5. 在一个该元件的插入位点,是转录起始位点--而不是CTCF结合位点--负责形成了新的接触结构域。而在另一位点,转录起始位点和CTCF结合位点分工施加了染色质近距离和远距离折叠的偏好。这个发现对于理解基因组上众多共定位(co-localized)的CTCF和TSS对于基因组架构的功效会有一定启发。
6. 一系列CRISPR基因剖析表明,在这个2 kb的元件中,指示染色质折叠的信息大部分编码于其中 ~100 bp汇集了CTCF结合位点和转录起始位点的区间内。两者其一或同时存在都会对附近基因组的cohesin结合水平有影响,暗示染色质区室化(compartmentalization)和环挤压(loop extrusion)两种模式也许并不是两种相互独立的过程。
有意思的是,这个2 kb边界元件在其原生位点(endogenous locus)被删除后,两个临界结构域并没有出现明显融合,而是边界被微调到了旁边另一个具有CTCF结合位点和转录起始位点的位置。这也进而意味着架构上的效应是由编码了指示基因组折叠信息的边界元件和其周围基因组环境(genomic context)共同决定的。
到底高等动物基因组的三维结构是如何在进化中形成新的结构域或是增添结构上的冗余(redundancy)或稳定性,还有待进一步研究。
总之,本文通过大量基因组编辑和多重架构测定展示了用短DNA片段来塑造三维基因组的可行性,并解开了结构域边界元件中CTCF和转录在不同基因组环境下对于染色质折叠的功效。
本文通讯作者为费城儿童医院与宾夕法尼亚大学医学院的Gerd A. Blobel MD, PhD, Frank E. Weise III Professor of Pediatrics, 第一作者和共同通讯作者为费城儿童医院及宾夕法尼亚大学医学院博士学生张帝。合作者包括宾州州立大学Ross C. Hardison, PhD的团队,以及宾夕法尼亚大学Jennifer Philips-Cremins, PhD的团队成员。(来源:科学网)

