我校动物遗传育种团队在基因组预测领域取得进展
南湖新闻网讯(通讯员 刘小磊)近日,国际学术期刊Genome Biology在线发表了题为“KAML: improving genomic prediction accuracy of complex traits using machine learning determined parameters”的研究论文。该研究提出了一种准确性高且计算高效的基因组预测方法,该方法利用机器学习的策略解析基因组和表型组大数据之间的隐藏关系,并根据表型的遗传复杂程度智能化选择最优预测模型来提高基因组预测的准确性。
基因组预测是指利用覆盖于基因组的高密度遗传标记对未知表型(或育种值)进行预测的技术。在动植物领域,利用该技术可对不同经济性状进行早期选择,保留优势个体,淘汰劣势个体,既能提高群体总体性能表现以获得丰厚的经济效应,还能极大降低饲养及表型测量成本;对于人类,基因组预测可根据遗传标记信息估计各类遗传疾病的患病风险,给人们的生活方式及饮食习惯提供针对性建议,保障人们的健康生活。预测准确性是基因组预测应用于实际的基本保证,而统计方法发挥至关重要的作用。线性混合模型(LMM)以其高效的计算效率优势成为目前基因组预测使用最广泛的方法,然而由于其简单的标记效应假设,预测准确性往往偏低,尤其对于受大效应基因影响的性状。另一类以贝叶斯(Bayes)理论为基础的方法,大多具有复杂的标记效应假设,模型灵活多变,能够适用于遗传构建从简单到复杂的性状,预测准确性往往高于LMM方法,然而其复杂的假设导致众多的未知待估超参,参数的求解过程无法并行运算,计算效率低下,尤其对于超高密度标记,预测一个性状可能需要数周甚至数月的时间,因此难以广泛应用于育种实践。
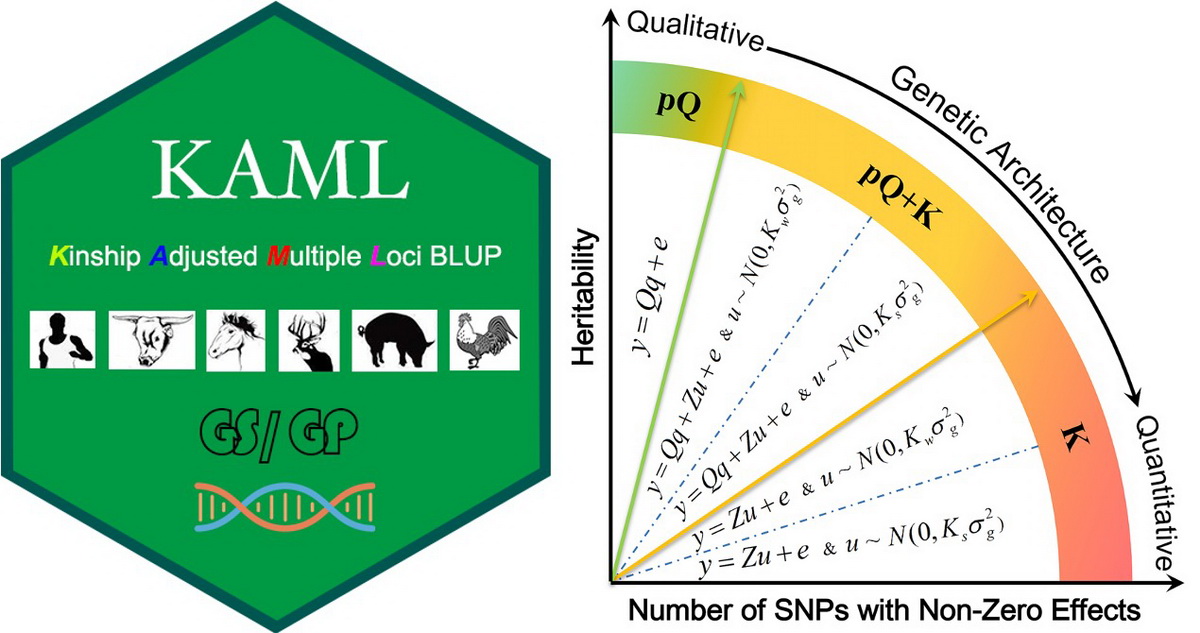
高计算效率的方法预测准确性较低,高预测准确性的方法计算效率较低。为了解决这一难题,该研究结合两类方法的特性,提出了一种准确性高且计算高效的新方法KAML。该方法利用高速可并行的机器学习策略解析性状的复杂程度,机器学习过程整合了交叉验证、多元回归、网格搜索以及二分求极值等方法,智能化选择最佳预测模型、最可靠的协变量QTN、最优的亲缘关系矩阵,多方面优化模型以达到最理想的预测准确性。研究结果显示,KAML具有与Bayes方法近似的准确性,在部分性状上甚至表现更好,显著超过LMM方法,计算效率高于Bayes方法30-100倍。同时,KAML可与动物育种中广泛应用的一步法(SS, Single Step)策略结合,研究结果显示SSKAML的预测准确性显著优于SSBLUP方法。另外,对于已被KAML分析过的性状,优化后的参数可直接用于新的群体预测,预测准确性几乎不变,计算效率等同于LMM方法。KAML和SSKAML可助力动植物基因组育种产业以及疾病风险预测等人类大健康产业的发展。
我校刘小磊副教授、李新云教授为文章共同通讯作者,博士生尹立林为论文第一作者,赵书红教授参与并指导了该项工作。同时,武汉理工大学袁晓辉教授、博士生张浩浩共同参与了该研究。上述研究工作得到了国家自然科学基金等项目的资助。
审核人:赵书红
KAML软件:https://github.com/YinLiLin/KAML
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-020-02052-w

