不只有A的poly(A)尾:A碱基纯度影响蛋白质翻译效率 | Genome Biology
论文标题:Impact of poly(A)-tail G-content on Arabidopsis PAB binding and their role in enhancing translational efficiency
作者:Taolan Zhao, Qing Huan, Jing Sun, Chunyan Liu, Xiuli Hou, Xiang Yu, Ian M. Silverman, Yi Zhang, Brian D. Gregory, Chun-Ming Liu, Wenfeng Qian & Xiaofeng Cao
发表时间:2019/09/03
微信链接:https://mp.weixin.qq.com/s/K6jlDcv1BJe2g52KglWf4g
poly(A)尾是真核生物mRNA最重要的特征之一,通常被认为由腺苷酸(A)的简单重复组成。近日,中国科学院遗传与发育生物学研究所、植物研究所与美国宾夕法尼亚大学开展合作,利用新型poly(A)尾高通量测序技术,揭示了拟南芥poly(A)尾介导的全新转录后调控机制——在poly(A)尾中散在分布的鸟苷酸(G)可通过抑制与poly(A)结合蛋白(PAB)的相互作用降低mRNA的翻译效率。该研究发表在Genome Biology 上。

poly(A)尾对真核生物mRNA具有关键的调控功能,是其稳定性的重要决定元件。尽管poly(A)尾非常重要,精确解读却非常困难。主要技术壁垒在于扩增简单串联的核苷酸序列会导致聚合酶滑动,从而造成测序结果的移码和乱码。近几年,一些针对mRNA尾的高通量测序技术逐步建立:例如,PAL-seq通过不同poly(A)长度样品的标准曲线估计待测定mRNA的poly(A)尾长度。Tail-seq则是针对二代测序的原始图像数据开发的算法,通过与标准品比较,推断poly(A)尾的长度及序列。人类细胞系中Tail-seq的结果显示poly(A)尾中存在非A核苷酸(G、U或者C),其中G所占比例最高。然而,由于难以进一步获得突变体,目前对于poly(A)尾中G的分子功能以及作用机制仍鲜有报道。
在本课题中,研究人员通过对全长poly(A)尾进行测序并发展下游生物信息学算法提取高质量测序信息,发现在模式植物拟南芥的poly(A)尾中也存在非A核苷酸,并且同样是G的含量最高。研究人员随后以拟南芥poly(A)结合蛋白家族核心成员AtPAB2、AtPAB4和AtPAB8为研究对象,构建了一系列重要的突变体。
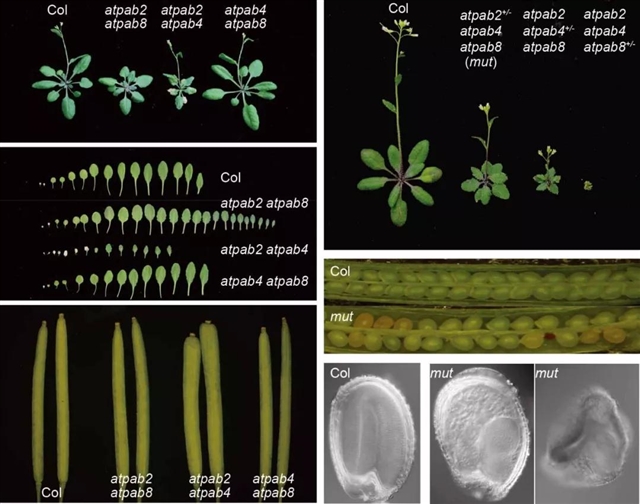
该研究通过进一步整合CLIP-seq、ribo-seq和mRNA稳定性检测等高通量实验技术,在全基因组水平对poly(A)尾及其结合蛋白PAB的分子生物学功能进行了系统性研究,得出以下结论:
1. AtPAB广泛结合植物体内mRNA的poly(A)尾,但对不同mRNA的结合效率存在明显差异;
2. 拟南芥中10%的poly(A)尾含有至少一个G,其含量在不同mRNA中的分布范围为0.8-28%;
3. AtPAB对不同mRNA的差异结合可以部分被其poly(A)尾中G含量的差异所解释——A的“纯度”越高,其与AtPAB的结合越强;
4. AtPAB对mRNA的结合可以提高mRNA的翻译效率;
5. 相应的,在atpab 突变体中,带有不含G的“纯poly(A)尾”的mRNA翻译效率降低更为明显;
6. atpab2 atpab4 atpab8 三突变体纯合致死,atpabs 的双突变体呈现多种发育异常表型,暗示了由poly(A)尾与其结合蛋白介导的翻译调节机制对植物的正常生长发育具有至关重要的调控作用。

综上所述,该研究充分展示了二代测序技术与生物信息学算法的结合在解析生物大分子调控过程中的强大优势。其结果阐明了植物poly(A)尾中的G含量可通过抑制PAB结合影响蛋白翻译效率,是对近代分子生物学中心法则的拓展与创新。该研究对探究其他物种中mRNA的转录后调控机理具有重要参考价值。

曹晓风研究员

钱文峰研究员
中国科学院遗传与发育生物学研究所曹晓风研究员和钱文峰研究员为论文的共同通讯作者;肇涛澜、郇庆、孙婧和刘春艳博士为共同第一作者;中科院植物所刘春明研究组、美国宾夕法尼亚大学Brian D. Gregory研究组参与了相关的研究工作。该研究得到国家自然科学基金委、科技部、中国科学院与植物基因组学国家重点实验室的资助。
摘要:
Background
Polyadenylation plays a key role in producing mature mRNAs in eukaryotes. It is widely believed that the poly(A)-binding proteins (PABs) uniformly bind to poly(A)-tailed mRNAs, regulating their stability and translational efficiency.
Results
We observe that the homozygous triple mutant of broadly expressed Arabidopsis thaliana PABs, AtPAB2, AtPAB4, and AtPAB8, is embryonic lethal. To understand the molecular basis, we characterize the RNA-binding landscape of these PABs. The AtPAB-binding efficiency varies over one order of magnitude among genes. To identify the sequences accounting for the variation, we perform poly(A)-seq that directly sequences the full-length poly(A) tails. More than 10% of poly(A) tails contain at least one guanosine (G); among them, the G-content varies from 0.8 to 28%. These guanosines frequently divide poly(A) tails into interspersed A-tracts and therefore cause the variation in the AtPAB-binding efficiency among genes. Ribo-seq and genome-wide RNA stability assays show that AtPAB-binding efficiency of a gene is positively correlated with translational efficiency rather than mRNA stability. Consistently, genes with stronger AtPAB binding exhibit a greater reduction in translational efficiency when AtPAB is depleted.
Conclusions
Our study provides a new mechanism that translational efficiency of a gene can be regulated through the G-content-dependent PAB binding, paving the way for a better understanding of poly(A) tail-associated regulation of gene expression.
(来源:科学网)

