Science:宏基因组学测序技术
宏基因组学技术(Metagenomic approaches)正快速拓宽我们对微生物代谢能力(microbial metabolic potential)的认识。
长期以来,对微生物(microorganism)功能开展的研究主要依赖的都是以在实验室里培养的单一物种(individual specie)为对象获得的研究成果。大约在10年前,科研人员们开始获得自然环境中存在的、非人工培养的细菌或古细菌(archaea)的基因组草图,这些基因组数据为科研人员们了解这些微生物在自然环境中的作用打开了一条新的渠道。这就是所谓的宏基因组学(metagenomics)技术,该技术的发展现在已经可以做到从多个不同的环境样本中快速、准确地获得环境微生物的基因组序列。这些成果有可能会彻底颠覆我们对生命之树结构,以及各个物种代谢能力的理解和认识。生物信息学的飞速发展也提供了另外一种便利,由于能够全面了解遗传信息和数据,所以能够快速地为这些宏基因组数据在医学诊断、农业、法医以及生物技术等应用领域里找到合适的位置和用途。
宏基因组学技术是一种不需要进行微生物培养的微生物研究方法,可以直接对取自环境里的微生物样品进行分析和研究。整个流程是先将样品里的DNA提取出来,进行测序,然后用计算机软件对测序结果进行分析。这种方法至少解决了两个非常重要的问题。首先,该技术能够让我们对大自然里99%的微生物(这些都还是没能在实验室里成功培养的微生物)进行分析和研究。其次,可以对整个大环境里的微生物进行研究。
由于这些DNA来自不同的物种,所以从宏基因组数据中找出每一个物种的基因组序列是一件非常复杂的工作。直到最近,科学家们才成功地从拥有丰富基因型、同时又相对简单的环境样品中获得了微生物的基因组序列。高通量DNA测序技术让我们有能力对生物物种相对没有那么丰富的环境样品进行分析,也能够确定拥有相当物种数量的环境。但是这会极大地增加数据分析工作的复杂程度和难度。新开发的计算机软件可以对数据进行合并,并且将分属于各个不同物种的基因组片段划归到相应物种的基因组里,这就是所谓的“框并法(binning)”。
2012年,Wrighton等人获得了49个细菌的基因组序列(这些序列的完整程度不一),这些细菌至少分属于5个不同的门(phyla),而在这之前我们对这些门类细菌的基因组信息几乎可以说是一无所知。Wrighton等人使用的就是框并法,这种方法将时间序贯丰度信息(time-series abundance information)与序列组成信息组合在一起。最近,Albertsen等人也使用多个样本的信息重建了31个基因组,这些基因组的平均完整性已经达到了80%。这一次使用的方法与分析人类婴儿肠道微生物组的方法类似。Albertsen等人也能够组装出TM7门类细菌(这是一大类目前还不能在实验室里培养的细菌)的完整基因组序列。科学家们也已经能够组装出在整个环境(比如海洋等)中只占1%的物种的完整基因组序列。最近还成功地获得了成年人肠道和大便微生物组的序列。这些成功的案例都表明,基于宏基因组的基因组序列捕获技术已经非常成熟,能够用于对高度复杂系统的科研工作当中。
不过序列准确性还是一个问题,影响了部分人对这种以宏基因组学技术为基础的测序技术的接受度。大家最关心的问题就是将来自不同物种的基因组序列给拼接到了一起。不过借助自动、或者人工的纠错机制是可以修正这种组装错误(Assembly error)的。而且我们还可以利用传统克隆测序策略里使用的验证机制来解决这个问题。很多时候,我们只需要将测得的序列与已经发表的、亲缘关系比较接近的物种的序列进行比对就可以明确测得序列的真实程度,也可以使用长片段、高质量的DNA测序技术进行验证。框并法是一种容易出错的方法,的确需要格外小心。使用多个不同来源的信息,尤其是在多个样本中物种丰度都非常独特的物种信息(内参)能够极大地降低错误发生的几率。
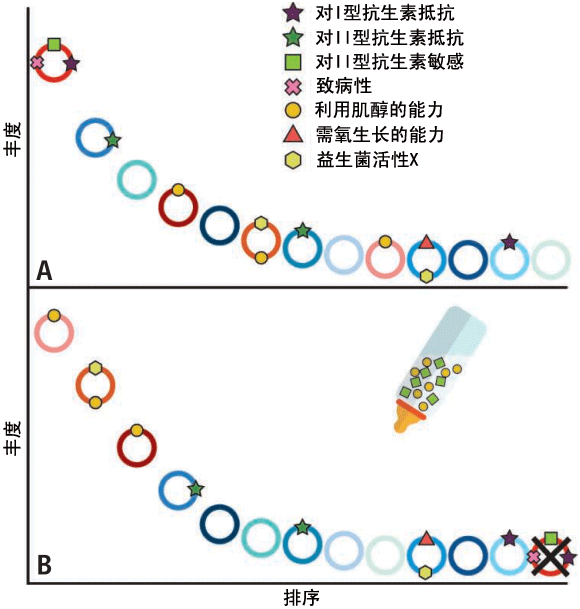
基于基因组信息的抗生素选择方案。能够掌握整体情况,明确其中每一种微生物功能的宏基因组学技术对临床工作也有一定的指导意义。如上图所示,根据环境中每种微生物的丰度从高至低进行排序。其中圆圈代表基因组。圆圈上的各种符号和标记代表各种性状,比如对抗生素的耐药性或者底物代谢能力等。在治疗之前(A),具有益生菌活性的微生物含量很少,其中大部分都是致病菌。根据宏基因组学分析之后给予相应的抗生素进行治疗,就能够大量消灭致病菌,使益生菌的丰度明显上升(B)。
除了宏基因组学技术之外,还有另外一种技术就是单细胞基因组测序技术,这也是一种不需要进行实验室培养的测序技术,只需要从环境样本中提取细胞并进行基因组测序就可以完成工作。最近,Rinke等人从多种不同的自然环境样本中都成功地分离得到了单细胞标本,并且成功地进行了DNA扩增和测序,获得了201个基因组(非完整基因组)。据估计,这些序列的平均完整程度达到了40%。据我们了解,到目前为止还没有哪一个单细胞基因组测序工作能够获得完整的基因组序列。这种单细胞基因组测序工作获得的序列基本上都是不完整的。Rinke等人开展的工作已经可以算是非常漂亮的工作了,他们获得的最完整的基因组序列(认为该序列的完整程度几乎接近100%)也是由10个片段拼接而成的,另外一个完整度达到99%的基因组序列更是由137个片段组成的。
用宏基因组学技术获得的基因组片段很多都是不完整的(这主要是因为样品不够),不过我们可以借助额外的测序工作加以弥补。可是对于单细胞基因组测序,再辅以其它测序似乎意义不太大。宏基因组学技术也不需要像单细胞测序技术那样费时费力,也不需要进行细胞分选,而且还能提供更多的信息。因为宏基因组学技术获得的是整体的信息。现在在基因组测序工作中使用的算法大部分都是株系特异性的(strain-specific),很有可能会测出多态性碱基。另外,如果被测细胞里存在基因插入或者缺失的情况,那么有一部分测序结果可能就只能与参考序列部分吻合了。如果科研人员对整体代谢能力、整体结构(population structure)、整体多样性或者进化动力学(evolutionary dynamics)等问题感兴趣,这些信息就对他们会非常有帮助。另外一方面,单细胞基因组测序技术也能够提供宏基因组学技术无法获得的信息,比如基因变异连锁信息(gene variant linkage information)等。
由于单细胞基因组测序技术和宏基因组学测序技术都获得了广泛的关注与认可,所以我们希望广大的科研人员能够使用定义非常明确的术语来描述基因组的完整性,以便于大家对这两种技术的优缺点进行更客观、更准确的评价。比如只有在经过仔细的检验之后,确认获得了一个完整的、连续的、没有错误或缺口的序列时,我们才可以称这条序列是一个完整的基因组序列。如果因为存在重复序列,或者其它原因导致基因组组装出现困难,得到了多条、而不是一条“基因组”序列,那么此时只能说得到了部分完整的基因组序列。根据Chain等人的工作,我们可以认为这种有多条序列的基因组序列是一个基因组草图。此时我们可以根据单拷贝基因名录来衡量该序列的完整性。由于单拷贝基因通常在整个基因组基因中所占的比例还不到10%,而且在基因组中的分布并不均匀,所以根据单拷贝基因也只能大概估计序列的完整程度。
因此还需要更强大的基因组草图完整性评估手段。使用在整个基因组中并非成簇存在的标志物基因(marker gene)也许是一个不错的办法。改进取样技术,获得更多不能培养的微生物的基因组样本,从中发现更多、更普遍存在的基因也会有所帮助。这些信息能够帮助科学家们从已知序列中发现更多的单拷贝基因。
随着测序技术不断发展,测序的速度、精度、信息丰富程度都有了大幅度的提升,高通量的宏基因组学技术也逐渐具备了给要求更快、更准确、特异性更高的整个测序诊断领域带来革命性改变的能力。比如如果发展到了个体微生物组(personal microbiomics)时代,我们就可以快速地对病原体群耐药基因进行测序和筛选,然后根据这些结果给出最合适的抗生素。根据宏基因组学数据还可以选择性地刺激目标微生物群,治疗肥胖症或慢性腹泻等疾病。更重要的是,如果掌握了生命之树中很多之前还没有被发现的基因组信息,将极大地丰富我们对生命和进化历程的认识和了解。

