【一天三弹】植物RNA是如何被编辑的?
南湖网讯(通讯员 洪思行)同在4月11日,Nature Plants以article形式在线发表了我校作物遗传改良国家重点实验室/生科院殷平教授团队关于植物RNA编辑复合体参与RNA编辑分子机制的最新研究成果。
类似于在一篇WORD文档中,会时常使用插入、删除、替换等手段来修改文字信息,被称为文档编辑。在植物遗传信息“天书”中,RNA编辑是指通过转录后特异位点核苷酸的插入、缺失及转换等加工修饰手段,对基因进行修饰和调控。在高等植物中,RNA编辑主要发生在线粒体和叶绿体中。RNA编辑异常影响植物细胞器的生物发生,导致植株生长缓慢,以及生殖发育障碍。
早在1989年研究人员就在植物中发现了RNA编辑的现象。2005年,第一个参与RNA编辑的蛋白因子被鉴定出来,发现它是一个PLS-type的PPR蛋白。随后,一系列的研究鉴定了多个PLS-type PPR蛋白家族作为反式因子参与RNA编辑。2012年,两个独立的研究组同时筛选到另一类RNA编辑因子——MORF蛋白家族(也称为RIP)。MORF突变体极大的降低线粒体和叶绿体RNA编辑的效率。
但是,这些蛋白因子是如何协同参与植物细胞器RNA编辑的分子机制并不清楚。
为了解决以上问题,该课题组首先试图通过结构生物学手段解析PLS-type PPR蛋白以及MORF蛋白的结构。由于大多数天然的PPR蛋白性质不好,很难拿到天然的PLS-type PPR蛋白进行结晶实验。该课题组通过将拟南芥中所有的PLS-type PPR蛋白进行序列保守性分析,巧妙的设计了一个含有9个重复序列包含3个PLS三联体重复的PLS-type PPR蛋白,命名为(PLS)3PPR。该蛋白可以进行异源可溶性表达,性质稳定。最终获得了PLS-type PPR蛋白的晶体结构,揭示了P,L,S这三类重复单元结构的差异性。
研究组同时对MORF家族蛋白成员进行结晶筛选,解析了MORF9蛋白的结构,发现其是一类新型的蛋白结构。研究人员通过生化实验证实(PLS)3PPR通过PLS三联体和MORF9相互作用,并解析了(PLS)3PPR - MORF9复合物晶体结构。结构显示L-type重复单元在介导蛋白相互作用过程中发挥关键的作用。有意思的是(PLS)3PPR - MORF9复合物比(PLS)3PPR单体有更强的RNA结合能力。结构比对发现,MORF9的结合驱使(PLS)3PPR 的L-type重复单元发生构型转变,使其更为紧凑,从而使得其第2位和35位的氨基酸距离更近,更有利于其对RNA碱基的结合。
基于以上发现,该研究组提出了MORF蛋白在RNA编辑中发挥功能的分子机制:在没有MORF蛋白存在的情况下,PLS-type PPR蛋白有较弱的靶标RNA结合能力;而当MORF蛋白存在时,可以引起PLS-type PPR蛋白构型发生改变,从而增强其对靶标RNA的结合能力。
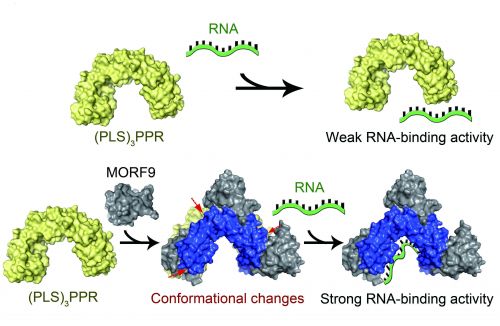
图示为叶绿体RNA编辑因子MORF9引起PLS-type PPR蛋白构型改变从而增强其对靶标RNA结合能力的分子机制模型
该研究由博士后闫俊杰、研究生张群霞作为论文共同第一作者,殷平教授作为论文通讯作者。中国科学院植物研究所林荣呈研究员为本研究提供了帮助。校级蛋白质平台为该研究的开展提供了强有力的支持。上海同步辐射光源(SSRF)为数据收集提供了帮助。该研究受到了科技部基金、华中农业大学科技自主创新基金和人才启动基金、以及中国博士后基金的资助。闫俊杰博士特别鸣谢学校优秀博士后持续职业发展基金项目的支持。
文章链接:http://www.nature.com/articles/nplants201737
审核人:殷平

