利用生物信息学发现基因组暗物质– 清华大学鲁志实验室非编码RNA软件系列介绍
非编码RNA(noncoding RNA)是基因组上不编码蛋白、在RNA水平发挥功能的一类基因,它们在生物体内行使着多种多样的重要功能。对于非编码RNA基因的鉴定、结构及功能研究是近年来生命科学领域研究的热点。清华大学鲁志实验室围绕非编码RNA,开发了一系列分析软件,现分享给大家,以供同行们学习交流。

I. RNAfinder软件系列: 利用机器学习发现基因组暗物质-新型非编码RNA基因
由清华大学鲁志实验室ncRNA Project (http://software.ncrnalab.org)开发的第一个软件系列RNAfinder发表了两个新的成果:RNAfeature和COME。

为了能够有效地鉴定非编码RNA基因,ncRNA project开发了两个配套的工具:RNAfeature (http://RNAfinder.ncrnalab.org/RNAfeature)和COME (http://RNAfinder.ncrnalab.org/COME)。RNAfeature整合并筛选了超过600套基因组学数据、表观基因组学数据、转录组学数据与多个进化学、生物物理学数值特征,得到了包括序列特征、结构特征、表达谱特征及表观遗传标记特征等10个共有特征可以跨物种跨种类描述非编码RNA。此外,COME可以将这些特征整合在一起并非常有效地鉴定新型非编码RNA。RNAfeature和COME各自发表于生物信息学代表性期刊Nucleic Acids Research上(2015和2016),被很多重要的基因组学工作采用,其中最具影响力的是国际重大研究计划ENCODE(人类基因组注释计划)。
II. RNAstructurome软件系列: 高通量预测RNA二级结构
由清华大学鲁志实验室ncRNA Project (http://software.ncrnalab.org) 开发的第二个软件系列RNAstructurome最近发表了一个新的算法RME,并配套了一个网络服务RNAex。

在中心法则中,RNA位于承上启下的位置。除了传递遗传信息,RNA还可以发挥结构介导的基因调控功能。但是,RNA结构目前还难以被准确解析。近年来,研究者们发明了一系列新的高通量结构测定技术,用于辅助RNA二级结构预测,在RNA功能生物学中取得了很大的突破。为有效利用这些新型数据,鲁志实验室首先开发了RME算法,实现了数据指导下的RNA二级结构预测,并搭建了RNAex在线服务器,提供了自动化的数据访问和软件使用。
RME算法(http://structure.ncrnalab.org/RME)基于统一的统计模型对高通量结构测定数据进行转换,而后将其加入到能量模型中,实现了整合两种信息的RNA二级结构预测。在数据区分度较好时,RME可以大幅提高RNA二级结构预测准确度。
RNAex服务器(http://structure.ncrnalab.org/RNAex)对目前主流的结构测定数据进行了统一处理,提供了4种模式物种的数据查询,和3种结构预测软件的在线使用,并将预测结构与功能信息进行统一可视化,使得用户可以快速有效的利用新型数据。
目前,RME算法和RNAex服务器各自发表于生物信息学代表性期刊Nucleic Acids Research上(2015和2016),并被OMICtools数据库收录,是RNA结构组学目录下的第一个代表性算法。
III. RNAtarget软件系列: 发现mRNA和非编码RNA的结合蛋白
由清华大学鲁志实验室ncRNA Project (http://software.ncrnalab.org)开发的第三个软件系列RNAtarget最近增添了2个新的工具:POSTAR和CLIPdb。
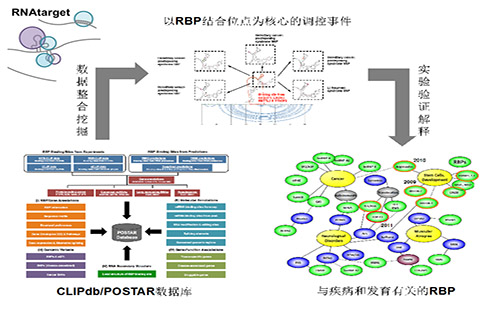
在真核细胞内,转录组与转录后调控有着密切的关联:转录本RNA从产生直至降解的过程中,总是被RNA结合蛋白(RBP)和其他RNA分子结合,这些调控因子参与几乎所有的转录后调控事件,包括RNA分子的剪接、加尾、亚细胞定位以及降解等。近年来,已经在包括癌症和神经退行性疾病等多种严重的人类疾病中发现了RBP及其参与的转录后调控的异常。然而,目前关于RBP参与转录后调控的数据库和计算工具还相当有限。因此,以大规模的CLIP-seq数据集为基础,整合其他高通量数据,研究人员构建了RBP转录后调控的整合型数据库CLIPdb和CLIP2-POSTAR。
CLIPdb(http://RNAtarget.ncrnalab.org/CLIPdb)是提供全转录组水平上高精度的RBP结合位点的数据库,共收集了来自四个物种(人类、小鼠、线虫与酵母)中400余套已发表的CLIP-seq数据集,涵盖了约120个RBP,使用统一的的计算方法在全转录组内鉴定RBP结合位点,并提供了方便的可视化和检索界面。
CLIPdb的第2版,POSTAR(http://RNAtarget.ncrnalab.org/POSTAR),提供了人类和小鼠转录组中目前已知最全面的来自实验证据和计算预测的RBP结合位点,涵盖了236个RBP。此外,利用大量的分子调控事件、基因组变异位点以及功能性基因等信息,POSTAR对RBP结合位点进行了详细的功能注释,为进一步深入研究RBP调控机制提供了重要的参考信息。
POSTAR和CLIPdb数据库已经发表于生物信息学权威期刊Nucleic AcidsResearch (2016) 和BMC Genomics (2015)和杂志,并且被Omic Tools工具库列为研究RBP调控功能的重要数据库之一。
本文来源于:测序中国
欢迎关注中科紫鑫人事招聘相关信息:http://www.ngscn.com/index.php/Job/employ

