中国科大发现一个长链非编码RNA在灵长类中进化出新功能
近日,中国科学技术大学教授单革课题组发现并命名了一个长链非编码RNA——5S-OT,并发现在灵长类中,5S-OT RNA获取了调控多个基因可变剪切的新功能。该研究成果发表在10月3日出版的《自然-结构和分子生物学》上。论文的共同第一作者为课题组的博士生胡珊珊和硕士生王小林。
从酵母到人类的各种真核细胞中,基因组DNA上的信息主要是由RNA聚合酶II及RNA聚合酶III读取并转录产生相应的RNA。这些RNA按照其是否会作为蛋白质翻译的模板,可以分为编码RNA(mRNA,即信使RNA)和非编码RNA。非编码RNA是一大类不编码蛋白质而在细胞中起着调控作用的RNA分子。而mRNA往往以大的pre-mRNA前体方式产生并随后进行剪切,产生能真正作为翻译蛋白质的mRNA模板。
研究人员发现,在高级灵长类和人类当中,5S-OT RNA获取了调控多个基因可变剪切的新功能。在高级灵长类和人类中,5S-OT中插入了一个反义Alu序列,并因此同时产生了一个多嘧啶(polypyrimidine-tract,Py)位点。Alu是一种灵长类特异的基因组短重复序列,而人类基因组DNA中约10%的序列为Alu的正向或反向序列。随后在人细胞中的研究发现,人类5S-OT RNA可以通过其Py位点招募参与pre-mRNA剪切的蛋白质U2AF65,进而通过5S-OT RNA中的反向Alu序列与靶标pre-mRNA当中的正向Alu序列的互补配对,将U2AF65蛋白带到受其调控的pre-mRNA上。非灵长类动物的5S-OT RNA不具有Py位点,不与U2AF65蛋白有相互作用,因而不参与调控相应物种中的mRNA可变剪切。
人们可以利用人类5S-OT RNA的这一调控机理,通过人工设计的、针对特定基因序列的“改装版”5S-OT来调控特定基因的剪切。很多人类疾病与基因剪切异常相关,未来有可能以这一发现为基础发展出生物技术来“纠正”基因的可变剪切。
上述研究得到了科技部、中科院、国家自然科学基金委等资助。
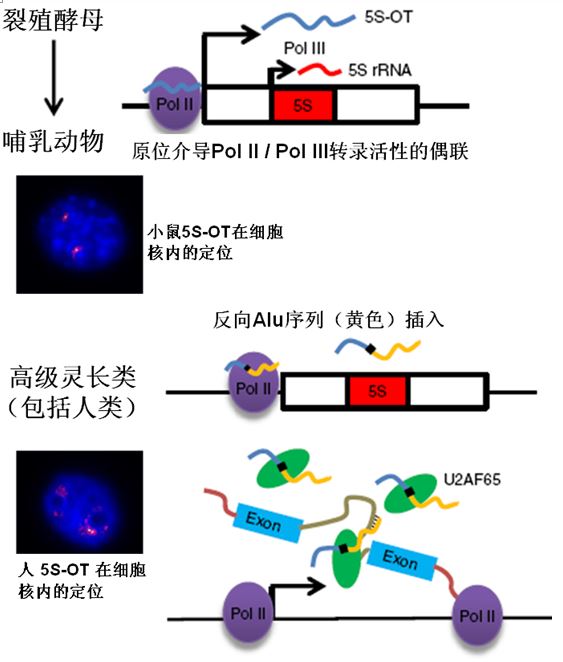
图:5S-OT RNA在从裂殖酵母到哺乳动物(包括小鼠、灵长类和人)细胞中均由RNA聚合酶II (Pol II)转录产生,并且在其产生处原位介导RNA聚合酶II 和RNA聚合酶III(Pol III)转录活性的协同。在高级灵长类及人细胞当中,5S-OT RNA进化出调控细胞核内一些基因剪切(即外显子Exon间的拼接)的功能。5S-OT RNA(照片中红色)定位于小鼠及人细胞核内(照片中蓝色)。

