“标准化”单细胞RNA测序数据可视化和解读的工具
加州大学伯克利分校的研究人员开发了一种软件FastProject,用于可视化和解读单细胞RNA测序数据,相关研究近日发表在《BMC Bioinformatic》上。

文章作者、加州大学伯克利分校电气工程和计算机科学系助理教授NirYosef说,“我的实验室对利用基因组工具研究转录的调控非常感兴趣。过去几年,我们研究了单细胞数据,当处理单细胞数据时,你面对的是海量的信息,它可能包含成千上万的细胞和基因。FastProject可以快速、直观地分析数据,并以一种非监督的方法寻找主题。”
FastProject解决了二维图中单细胞RNA测序数据可视化相关的三个问题:1. 选择一种合适的数据投影方式;2. 创建投影方式后,如何理解投影数据的生物学意义,例如观察到的细胞形态与哪种表型相对应;3. 混杂因素的控制,例如基因捕获率的差异,它可以导致单细胞RNA数据难以解读。
Yosef说,“FastProject软件解决了长久以来都没有得到处理的问题——数据投影的解读。几乎所有的RNA测序文章中都可以看到细胞的二维图,人们用各种方法解读这些图,每篇文章都不相同。”FastProject为数据投影的解读提供了一种“标准方法”,尽可能做到无偏好性。
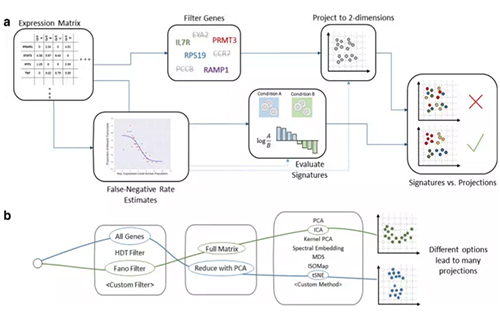
The FastProject pipeline
FastProject软件的工作原理
具体来说,FastProject使用了不同的线性和非线性投影方法使数据可视化。Yosef解释道,“通过这种方法,你可以对所有的二维投影有大致了解,并且知道每个投影代表什么,以一种简单的方式来进行观察和比较。”这个软件将基因标签信息(具有共同功能的基因的集合)整合到创建的投影中,以便用户在它们可能代表的生物学过程的背景下探讨数据点。该软件包含对细胞-基因标签评分的工具,以降低遗漏转录本的影响,以及对基因标签-投影匹配度排名的工具,以突出数据中具有意义的关联。
FastProject的输入文件包括,制表符分隔格式的表达矩阵;来自标准数据库的基因标签,例如Broad研究所的分子标签数据库(Molecular Signatures Database)、GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Genes and Genomes)。用户还可以创建自己的基因标签,来代表感兴趣的特定表型,并将这些作为输入文件。
首先,该软件评估数据集中的假阴性,使用不同的标准过滤掉在低于阈值细胞数目中出现的基因,默认阈值是细胞数的20%。接下来,该软件利用11种不同的投影方式为每个细胞生成二维坐标图,并利用基因标签数据库中的信息对每对细胞-标签进行评分,使用随机测试对统计学上有显著意义的投影-标签相关性进行识别和排名。
该软件产生76种可能的投影以及相关的功能注释,并以一份简单易懂的报告提供这些信息。同时它还能生成结果的文本文件,可帮助用户更加简单地研究与二维位置高度地相关的不同通路间的关联,还能突出数据中的一些新关联。
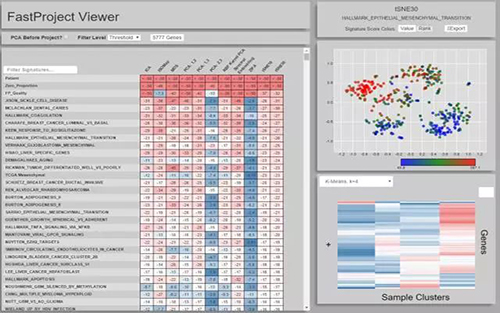
FastProject Output Report
Yosef说,“常用的解读单细胞数据的流程是,对细胞聚类,然后对不同的类群进行差异表达分析,以解读其生物学意义。而FastProject提供了另外一种选择,既适用于可从数据中清晰推测出聚类或亚群的情况,也适用于不能明确划分亚群的情况。”
该软件的分析结果是准确的。加州大学伯克利分校的研究人员利用FastProject对来自GEO(Gene Expression Omnibus)数据库的5名胶质母细胞瘤患者的430份肿瘤细胞进行分析,结果显示该软件可以根据细胞各自的供体对细胞进行正确的分层,与数据库已发布的结果相一致。
Yosef说,“利用FastProject软件我们可以得出相同或类似的结果。与那些需要更多操作和劳力的方法相比,这种工具更加全面和系统。”
参考文献:
FastProject: a tool for low-dimensionalanalysis of single-cell RNA-Seq data. DOI: 10.1186/s12859-016-1176-5.
本文来源于:测序中国
欢迎关注中科紫鑫人事招聘相关信息:http://www.ngscn.com/index.php/Job/employ

