PNAS:农科院林敏研究组揭示新型非编码RNA的固氮调控和协同进化
近日,国际著名学术期刊《美国国家科学院院刊》杂志在线发表了中国农业科学院生物技术研究所微生物功能基因组团队林敏研究组在解析生物固氮机制的研究新进展。论文题为“The novel regulatory ncRNA, NfiS, optimizes nitrogen fixation via base pairing with the nitrogenase gene nifK mRNA inPseudomonas stutzeri A1501”战嵛华和燕永亮两位博士为该论文共同第一作者,林敏研究员为通讯作者。
研究发现分离自我国南方水稻根际土壤的施氏假单胞菌A1501基因组携带一个固氮基因岛,其表达受两个进化来源不同的网络调节系统的精细控制,而非编码rna在最佳固氮调节中发挥了重要作用。分子生物学证据表明,固氮酶基因nifK招募了感受逆境信号的非编码调控因子NfiS,并且经过长期的协同进化,使其mRNA稳定性或翻译活性受到NfiS高效而精细的调控。该非编码RNA在抗逆与固氮途径间建立一种确保高效固氮的新的调控偶联机制,是目前报道的第一个直接参与固氮调控的非编码RNA,可望成为一个生物固氮智能调控的候选元件。
据了解,作物根际联合固氮菌在长期进化中形成了一套复杂而精细的基因表达网络调控系统,以适应复杂而多变的外界环境。目前,联合固氮菌如何感应环境信号的分子机制尚不十分清楚,此外非编码RNA是否参与固氮调节尚无直接试验证据。本研究发现了一个直接参与固氮基因表达调控的非编码RNA NfiS,通过实验证明其感应外界逆境信号和固氮信号,与不同目标基因mRNA直接或间接的相互作用,增强固氮菌的逆境抗性与固氮酶活。同时,研究进一步揭示了固氮施氏假单胞菌进化过程中发生的两次重大进化事件:(1)通过基因岛转移获得固氮能力;(2)通过招募NfiS精细调节固氮使其酶活最佳化。这两次进化事件赋予了水稻根际固氮菌A1501更强的环境适应能力。NfiS的发现及其调节机制的揭示为打破碳氮限制因子对联合固氮田间应用的制约,增强联合固氮效率,实现非豆科农作物节肥稳产增效奠定了重要理论基础。
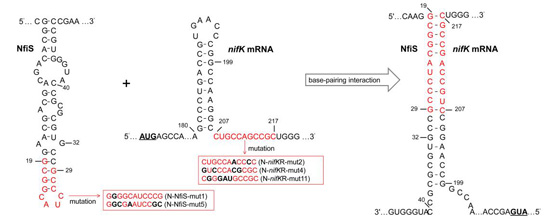
图1 NfiS与固氮酶结构基因nifK mRNA直接相互作用。NfiS的颈环结构参与固氮酶活的调节,影响nifKmRNA稳定性或翻译活性。
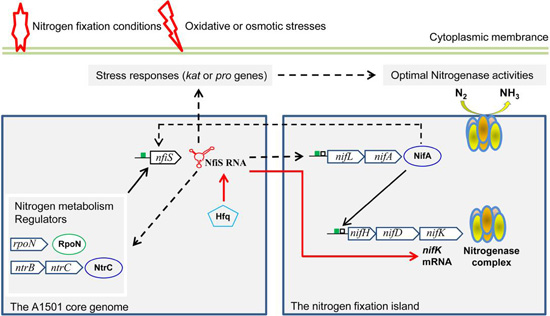
图2 NfiS介导的最佳固氮网络调控模式。NfiS在抗逆与固氮途径之间建立一种全新的调控偶联,确保高效固氮。
原文链接:
The novel regulatory ncRNA, NfiS, optimizes nitrogen fixation via base pairing with the nitrogenase gene nifK mRNA inPseudomonas stutzeri A1501
原文摘要:
Unlike most Pseudomonas, the root-associated bacterium Pseudomonas stutzeri A1501 fixes nitrogen after the horizontal acquisition of a nitrogen-fixing (nif) island. A genome-wide search for small noncoding RNAs (ncRNAs) in P. stutzeri A1501 identified the novel P. stutzeri-specific ncRNA NfiS in the core genome, whose synthesis was significantly induced under nitrogen fixation or sorbitol stress conditions. The expression of NfiS was RNA chaperone Hfq-dependent and activated by the sigma factor RpoN/global nitrogen activator NtrC/nif-specific activator NifA regulatory cascade. The nfiS-deficient mutant displayed reduced nitrogenase activity, as well as increased sensitivity to multiple stresses, such as osmotic and oxidative stresses. Secondary structure prediction and complementation studies confirmed that a stem-loop structure was essential for NfiS to regulate the nitrogenase gene nifK mRNA synthesis and thus nitrogenase activity. Microscale thermophoresis and physiological analysis showed that NfiS directly pairs with nifK mRNA and ultimately enhances nitrogenase activity by increasing the translation efficiency and the half-life of nifKmRNA. Our data also suggest structural and functional divergence of NfiS evolution in diazotrophic and nondiazotrophic backgrounds. It is proposed that NfiS was recruited by nifK mRNA as a novel regulator to integrate the horizontally acquired nif island into host global networks.
作者:林敏

