北京基因组所升级开发进化树可视化和注释...
发表日期:2016-05-10 02:33PM 阅览次数:
进化树是反映物种之间进化关系的常用可视化工具,用来表示不同物种亲缘关系的远近。为方便研究人员快速解读进化树中所包含的信息,中国科学院北京基因组研究所基因组科学与信息重点实验室胡松年组于2012年开发了进化树可视化和注释工具Evolview。该软件支持多种常用进化树格式(如newick, nexus, nhx等),并支持以多种方式对进化树进行注释,比如将注释信息显示为“饼图”、“条形图”等。
近期,在Evolview原有注释功能基础上,该团队添加了7种新的注释,包括点阵图、热图、分组标签等。此外,Evolview还提供5个个性化的展示案例,每个展示案例综合利用了多种新的注释工具对进化树进行解读,方便研究人员熟悉新的注释工具,赋予进化树更多的生物学意义。该成果于4月30日在Nucleic Acids Research 杂志在线发表,基因组科学与信息重点实验室研究员胡松年与客座研究员陈卫华是该篇文章的共同通讯作者。
自2012年发表以来,Evolview受到众多用户的欢迎。目前为止,共有2500个用户上传了12000个进化树和8600个注释文件,月活跃用户、上传数据量和引用次数都在持续增加。目前每月大约有200名活跃用户,上传550个进化树和450个注释文件。该工具为科研人员进行进化生物学、流行病学以及物种分类研究提供了极大便利。
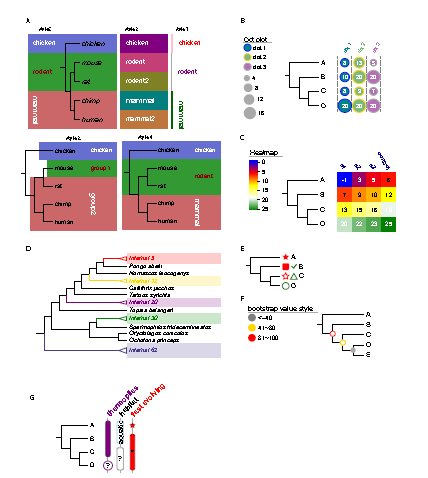
Evolview新增的7种注释工具示意图

