Plant Physiol:中科院昆明植物所刘爱忠研究组发表双子叶胚乳型种子基因组DNA甲基化研究文章
2016年4月28日,国际植物学顶级期刊《Plant Physiology》杂志上在线发表中国科学院昆明植物研究所刘爱忠研究组最新双子叶胚乳型种子基因组DNA甲基化研究进展文章,研究成果题为“Genomic DNA methylation analyses reveal the distinct profiles in castor bean seeds with persistent endosperms”。博士研究生许巍为论文第一作者,刘爱忠研究员为论文通讯作者。
DNA甲基化是一种非常重要的表观修饰因子,参与异染色质的形成,转座子的沉默,基因表达的调控以及印迹基因的发生。在植物中,DNA甲基化主要发生在CG,CHG和CHH序列上(其中H代表非G的碱基类型),分别由MET1,CMT3和RNA指导的DRM2甲基转移酶来维持;同时,开花植物中还存在DNA的主动去甲基化过程,由DNA糖基化酶通过剪切修复机制实现。目前在植物中主要发现三类DNA糖基化酶即:DME,ROS1,DML2和3。因此,基因组最终的甲基化水平是由DNA甲基转移酶和去甲基化酶的活动共同决定。越来越多的证据显示DNA甲基化对种子发育和储存物质的生物合成具有重要的调控作用。
虽然研究发现单、双子叶植物基因组甲基化式样分化明显,而且在拟南芥、水稻和玉米的种子中,胚乳基因组(相对于胚基因组)表现出广泛的低甲基化,但是由于双子叶模式植物拟南芥的胚乳组织只存在种子发育早期,随着种子发育逐渐消失,长期以来在双子叶植物胚乳中研究基因组低甲基化的生物学意义受到限制。蓖麻种子是典型的双子叶胚乳型种子,是种子生物学研究的模式材料,也为研究双子叶植物胚乳基因组低甲基化的生物学意义提供了理想的材料。
研究人员对蓖麻胚和胚乳组织的基因组DNA甲基化,及其生物学意义进行了深入研究,揭示了蓖麻种子基因组中CHH甲基化是主要的甲基化形式,和其它种子植物如拟南芥、水稻和玉米相比,蓖麻种子DNA甲基化图谱显著不同,暗示了植物种子基因组DNA甲基化式样的不保守性。特别是,和胚乳基因组CG和CHG低甲基化相比,CHH 的甲基化没有展示显著的低甲基化。进一步,结合DNA甲基化相关基因的表达以及调控DNA甲基化的small RNA的表达谱分析,研究揭示了蓖麻胚乳CG和CHG甲基化水平的下降,与MET1和CMT1甲基转移酶的表达抑制以及DME去甲基化酶的表达激活有关。而且胚乳中CG与CHG甲基化水平的下降与胚乳偏爱性基因的表达密切相关,而且研究发现这些基因广泛地参与了胚乳的发育过程。同时,研究发现胚乳中高丰度的CHH甲基化与24-sirnas介导的RdDM途径以及DRM3甲基化转移酶的表达激活有关。该研究不仅崭新地揭示了双子叶胚乳型种子基因组甲基化的规律,而且为研究植物种子基因组甲基化的生物学意义提供了重要依据。
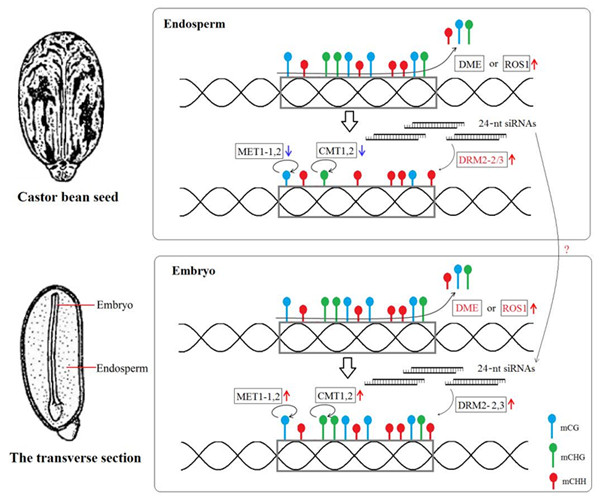
蓖麻胚和胚乳基因组DNA甲基化调控的模式
原文链接:
Genomic DNA methylation analyses reveal the distinct profiles in castor bean seeds with persistent endosperms
原文摘要:
Investigations of genomic DNA methylation in seeds have been restricted to a few model plants. The endosperm genomic DNA hypo-methylation has been identified in angiosperm, but it is difficult to dissect the mechanism how this hypo-methylation is established and maintained because endosperm is ephemeral and disappears with seed development in most of dicots. Castor bean (Ricinus communis), unlike Arabidopsis, its endosperm is persistent throughout seed development, providing an excellent model to dissect the mechanism of endosperm genomic hypo-methylation in dicots. We characterized the DNA methylation-related genes encoding DNA methyltransferases and demethylases, and analyzed their expression profiles in different tissues. We examined the genomic methylation including CG, CHG and CHH contexts in endosperm and embryo tissues using bisulfite sequencing and revealed that the CHH methylation extent in endosperm and embryo was, unexpectedly, substantially higher than known plants, irrespective of the CHH percentage in their genomes. In particular, we found that the endosperm exhibited a global reduction in the CG and CHG methylation extents relative to the embryo, markedly switching the global gene expression. However, the CHH methylation occurring in endosperm did not exhibit a significant reduction. Combining with the expression of 24-nt siRNAs mapped within TE regions and genes involved in the RdDM pathway, we demonstrated that the 24-nt siRNAs played a critical role in maintaining CHH methylation and repressing the activation of TEs in endosperm persistent development. This study discovered a novel genomic DNA methylation pattern and proposed the potential mechanism occurring in dicot seeds with persistent endosperm.
作者:刘爱忠

