计算生物学所科研人员开发用于准确快速估算遗传重组率的新算法
3月30日,国际学术期刊G3-Genes Genomes Genetics在线发表了中国科学院上海生命科学研究院计算生物学研究所李海鹏研究组的最新研究论文“New software for the fast estimation of population recombination rates (FastEPRR) in the genomic era”。该工作开发了一个快速、准确估计遗传重组率的算法及相关软件包FastEPRR,其遗传重组率估值与目前国际上最为常用的LDhat一样精确,但是比LDhat快了超过30万倍。
遗传重组是在减数分裂过程中发生的一个极为重要的生物学现象,为生命进化提供了丰富的素材。遗传重组率对于连锁不平衡研究,以及全基因组关联分析准确定位致病突变研究等都是极为关键的,因此遗传重组长期以来是生命科学领域的一个研究热点(目前在PubMed上查询到的与遗传重组相关的文献多达27万余篇)。目前一般采用对配子、或者父本-子本进行测序,对遗传重组事件进行直接计数,或者通过群体遗传学的方法建立全基因组的遗传重组图谱。然而由于多数生物的遗传重组率水平极低,所以前者的应用受到了很大局限。而基于群体遗传学理论估计遗传重组率的现有方法极为耗时(Griffiths and Marjoram 1996; Fearnhead and Donnelly 2001),即使在不断优化后也依然如此(Hudson 2001; McVean, et al. 2002)。随着新一代测序技术的不断迅猛发展,数据产生的速度已经远远超出了数据分析的能力,且样本量越来越大,因此提出新的高效算法迫在眉睫!
针对此问题,李海鹏研究组前期已提出了一个新的、基于回归分析的理论方法(Lin et al, 2013, Genetics)。以此为基础,该研究对这一理论方法进行了多方面扩展并实现在FastEPRR软件包中。针对单个DNA片段,当样本量为100个个体时,FastEPRR所需计算时间比牛津大学开发的、目前国际上使用最为普遍的LDhat快30万倍以上(少于2分钟 vs 499天),并且精确度达到了与后者完全一样的水平。在普通个人电脑上(单核)使用FastEPRR在3天之内即可顺利完成千人基因组数据的分析,如果使用多核CPU或者计算机集群,其计算时间会显著降低。而随着样本量的增加,LDhat所需计算时间却接近指数级增加。因此,FastEPRR很可能是目前唯一一个可以顺利处理万人基因组级、甚至十万人基因组级数据的分析工具,可以快捷准确地建立遗传重组图谱。可以预期这一创新性的新方法和软件分析平台将极大地促进相关领域的研究工作。
该研究主要由李海鹏研究组的博士研究生高峰完成,得到了中国科学院、国家自然科学基金委、科技部的经费支持。(计算生物学所)
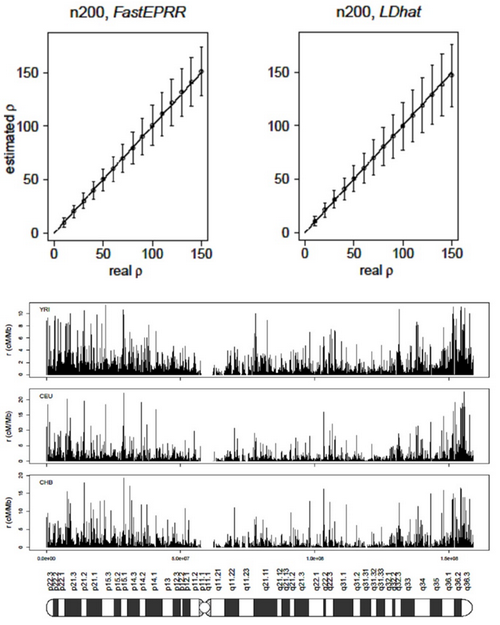
FastEPRR与LDhat遗传重组率(ρ)估值精确度的比较(上方)
FastEPRR对三个人类群体(YRI, CEU 和CHB) 7号染色体遗传重组率的估值及其遗传重组热点(下方)
相关附件
